El género Brettanomyces, particularmente Brettanomyces bruxellensis, ha sido ampliamente estudiado debido a su notable diversidad genética y su capacidad para adaptarse a una variedad de entornos industriales como la elaboración de vino, cerveza, kombucha y bioetanol (Schifferdecker et al., 2014). Su presencia puede cambiar completamente las propiedades organolépticas del producto, debido principalmente a la producción de metabolitos secundarios característicos. Así pues, Brettanomyces está asociada a procesos de fermentación en los que puede considerarse como un agente perjudicial (por ejemplo, elaboración de vino, sidra o producción de bioetanol) o como un microorganismo beneficioso (por ejemplo, elaboración de cervezas específicas o kombucha). Esta levadura es conocida por su capacidad de prosperar en diferentes nichos industriales gracias a su plasticidad genómica, hibridación, variación en la ploidía y otros mecanismos evolutivos (Curtin et al., 2012). Este artículo describe un análisis detallado de la diversidad genética de Brettanomyces, destacando su variación en la ploidía, plasticidad genómica, diversidad fenotípica, eventos de hibridación y las implicaciones productivas de estos rasgos.
Breve historia del género Dekkera/Brettanomyces
En 1904 Hjelte Clausen aisló por primera vez levaduras de la especie Brettanomyces en el laboratorio de investigación de Carlsberg, donde se la consideró responsable de realizar la fermentación secundaria y el desarrollo de los sabores característicos de las mejores cervezas inglesas (Claussen, 1904). Su nombre viene de brettano por su procedencia de Bretaña y myces del griego myketes o fungus en latín. Curiosamente, este aislamiento inicial de Brettanomyces dio como resultado el primer microorganismo patentado de la historia (UK patent GB190328184). La primera investigación sistemática de las levaduras Brettanomyces fue realizada y publicada por Mathieu Custers en 1940, quien caracterizó 17 cepas diferentes aisladas de cervezas inglesas y belgas (Custers, 1940). Más tarde Peynaud y Domercq describieron la presencia de aislados de Brettanomyces en uvas y vinos, como organismos contaminantes (Peynaud & Domercq, 1956).
Inicialmente, la clasificación de esta especie se basaba únicamente en unas pocas variantes que se reproducían asexualmente (anamórficas) (Custers, 1940). Sin embargo, unas décadas más tarde, se observó la formación de ascosporas en algunas cepas y el género Dekkera se introdujo en la taxonomía como la contraparte teleomórfica (sexual) de Brettanomyces (Van der Walt, 1984). Por lo tanto, en las clasificaciones actuales, las levaduras que pertenecen al género Brettanomyces no forman esporas (anamorfas), mientras que el nombre del género Dekkera describe las variantes formadoras de esporas (teleomorfas) de la levadura, aunque se utilizan a menudo como sinónimos. Actualmente, se describen cinco especies de Brettanomyces/Dekkera, basadas en el análisis molecular de los géneros: los anamorfos B. bruxellensis, B. anomalus, B. custersianus, B. naardenensis y B. nanus, existiendo teleomorfos para las dos primeras especies, D. bruxellensis y D. anómala (Steensels et al., 2015). Entre estas, la especie mayoritaria en procesos fermentativos y la más estudiada es B. bruxellensis.
Variación en la ploidía y plasticidad genómica
Uno de los aspectos más llamativos de B. bruxellensis es su variación en la ploidía, que juega un papel crucial en su diversidad genética y adaptabilidad (Borneman et al., 2014). Las cepas B. bruxellensis presentan diploides, autotriploides y alotetraploides, lo que les permite adaptarse a entornos productivos específicos, tales como la elaboración de cerveza o bioetanol (Hellborg & Piškur, 2009). Por ejemplo, se ha encontrado que las cepas triploides son comunes en la elaboración de cerveza debido a su mayor resistencia a los estreses de fermentación (Gallone et al., 2018). Recientemente, Harrouard et al. (2023) representaron la diversidad genética de las subpoblaciones de B. bruxellensis y el nivel de ploidía (Figura 1) basándose en una serie de estudios previos (Avramova, Vallet-Courbin, et al., 2018; Eberlein et al., 2021; Fournier et al., 2017; Gounot et al., 2020).
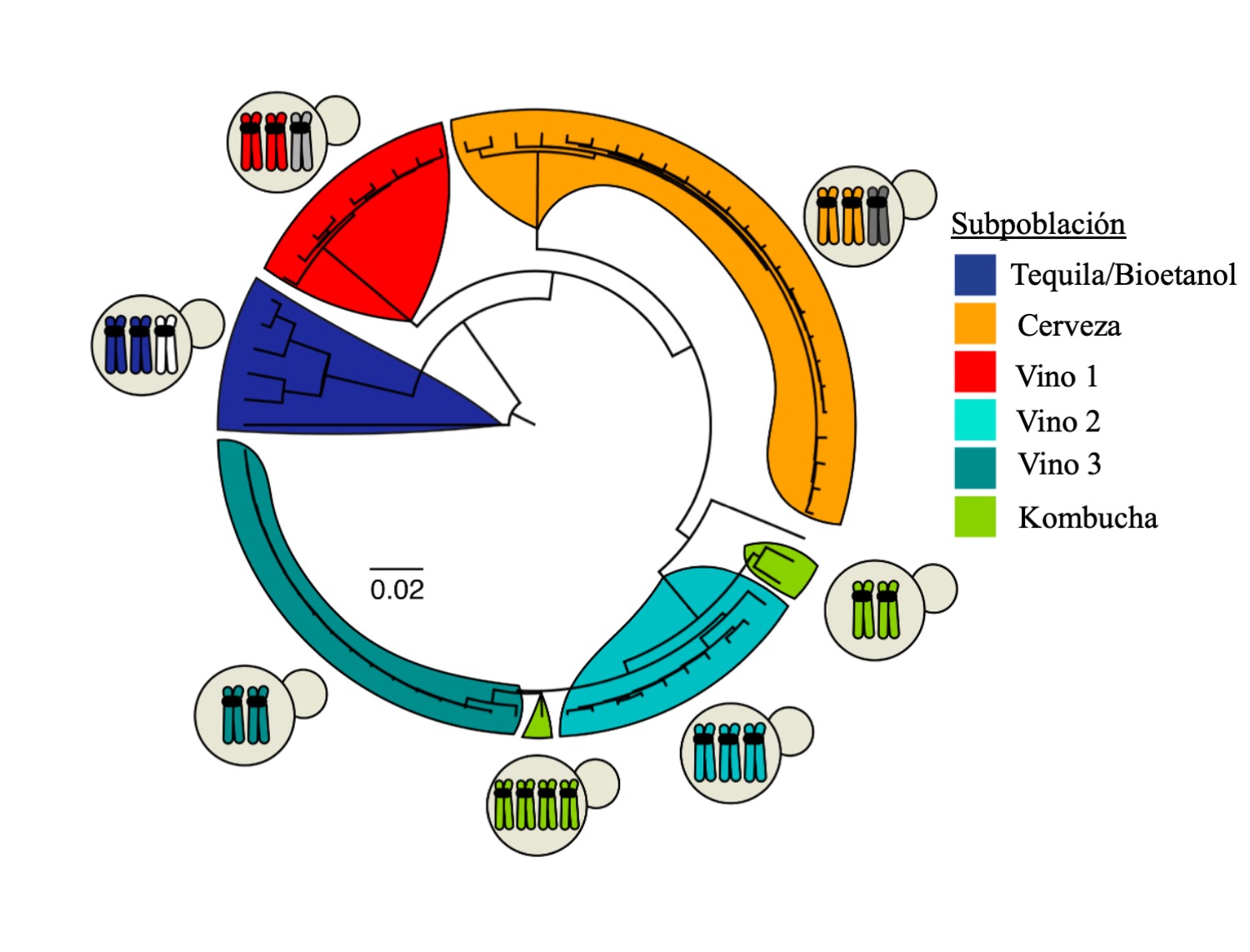
Además de la ploidía, la plasticidad genómica de Brettanomyces juega un papel esencial en su capacidad para adaptarse. Esta levadura muestra variaciones estructurales en su genoma, incluidas reorganizaciones cromosómicas, duplicaciones y deleciones que le permiten sobrevivir en condiciones de fermentación adversas, como altas concentraciones de etanol y bajos niveles de nutrientes (Curtin et al., 2007). Un estudio reveló que las reorganizaciones genómicas en B. bruxellensis mejoran su versatilidad metabólica, permitiéndole utilizar fuentes de carbono como celobiosa y lactosa, que son inaccesibles para otras levaduras (Piškur et al., 2012).
Alta variabilidad genética inter e intraespecífica
En 2007, Woolfit et al. publicaron el primer estudio del genoma de la cepa B. bruxellienses CBS2499, responsable del deterioro de vinos franceses, y proporcionaron una primera visión de la naturaleza peculiar del genoma B. bruxellensis. La obtención de genomas completos de alta calidad de las cinco especies de Brettanomyces reveló dos grupos genéticos distintos: uno formado por B. nanus y B. naardenensis, y el otro por B. bruxellensis, B. anomalus y B. custersianus (Roach & Borneman, 2020). La similitud promedio entre los genomas de estas especies varió entre el 60,6% y el 77,1%, mostrando que las especies de Brettanomyces están relativamente distantes entre sí. En comparación, las especies más distantes en el género Saccharomyces muestran una identidad del 79,9%. El estudio del genoma de estas especies de Brettanomyces demostró la presencia de expansiones genéticas relacionadas con la fermentación y la asimilación de nitrógeno, así como 12 eventos de transferencia horizontal de genes que podrían explicar la capacidad de B. bruxellensis y B. anomalus para utilizar sacarosa (Roach & Borneman, 2020). Estas características genéticas también se encontraron en poblaciones de S. cerevisiae tras su domesticación, lo que sugiere que Brettanomyces puede ser un modelo útil para estudiar la adaptación de las levaduras a ambientes fermentativos.
B. bruxellensis también es conocida por su alta variabilidad genética intraespecífica, que ha sido revelada a través de estudios de asociación del genoma completo y tipificación de secuencias multilocus (Albertin et al., 2014). Estos estudios han identificado linajes genéticos distintos que se correlacionan con nichos industriales específicos. Recientemente, Lebleux et al. (2021), utilizando un método adaptado de PCR con ADN polimórfico amplificado al azar (RAPD) y tres cebadores diferentes en aislados de B. bruxellensis, han encontrado una alta correlación entre los resultados obtenidos con uno de estos cebadores y un análisis previo de microsatélites (Avramova, Vallet-Courbin, et al., 2018), lo que permitió agrupar los aislados en cuatro grupos genéticos de forma más rápida y económica. Además, se investigó la correlación entre los grupos genéticos y el polimorfismo celular mediante imágenes microscópicas analizadas con deep learning, pudiéndose predecir el grupo genético con una precisión del 96,6% mediante una red neuronal convolucional (Lebleux et al., 2021).
Perspectivas genómicas de los estudios de población
Los estudios genómicos de poblaciones han proporcionado información sobre la adaptación de Brettanomyces a entornos productivos específicos. Se han identificado grupos genéticos de B. bruxellensis adaptados a diferentes nichos, lo que revela una clara diferenciación entre las poblaciones que habitan en entornos de elaboración de vino y cerveza (Roach & Borneman, 2020). Oro et al. (2019) comprobaron que las cepas aisladas tanto del viñedo como de la bodega pertenecían a biotipos comunes y dominantes, lo que sugiere que el viñedo puede ser la fuente de contaminación de B. bruxellensis en la bodega. Además, las cepas aisladas de la elaboración de vino han desarrollado una mayor capacidad para entrar en un estado viable pero no cultivable (VBNC); un mecanismo de supervivencia que les permite persistir en condiciones difíciles (Goddard, 2008). No obstante, B. bruxellensis es difícil de aislar de entornos naturales debido a su baja difusión, poca presencia en la superficie de la uva, baja capacidad competitiva, crecimiento lento y su estado VBNC, incluso cuando se utilizan medios selectivos para su aislamiento. Un estudio sobre las cepas vinícolas de Brettanomyces sugirió que la capacidad de entrar en el estado VBNC está relacionada con su adaptación a los estreses de fermentación, como el uso de agentes antimicrobianos y altas concentraciones de etanol (Serpaggi et al., 2012).
Aunque tradicionalmente se considera un organismo de deterioro en fermentaciones de producción de bioetanol, se ha descubierto una cepa B. bruxellensis capaz de competir exitosamente con S. cerevisiae sin afectar la producción de etanol. Además, B. bruxellensis tiene características como la capacidad de consumir nitrato y dextrinas, lo que podría expandir el rango de sustratos utilizados en la producción industrial de bioetanol (Reis et al., 2014).
Este tipo de mecanismos de supervivencia refuerza la idea de que la presión antropogénica en los entornos industriales ha sido un motor clave en la evolución y diversificación de Brettanomyces.
Hibridación y flujo génico
La hibridación ha sido un proceso evolutivo importante para Brettanomyces, ya que ha dado lugar a nuevas cepas con características mejoradas. Se ha documentado la existencia de cepas alotetraploides que surgieron de eventos de hibridación entre Brettanomyces y otras especies de levaduras (Steensels et al., 2015). Estas cepas híbridas han demostrado tener mayor resistencia al estrés y mayor versatilidad metabólica (Piškur et al., 2012).
Los eventos de hibridación entre especies no son raros en los hongos y también se observan en Saccharomyces sensu estricto. Por ejemplo, la levadura de cerveza S. pastorianus es una especie híbrida de S. cerevisiae y S. eubayanus (Libkind et al., 2011). Los genomas híbridos recién formados tienden a ser muy inestables como se ha mostrado anteriormente para híbridos de Saccharomyces (Antunovics et al., 2005). Los mecanismos que impulsan la estabilización del genoma podrían explicar la extrema variabilidad cariotípica observada en B. bruxellensis.
El flujo génico entre las poblaciones de Brettanomyces también contribuye a su diversidad genética. La transferencia de cepas de levaduras entre diferentes entornos industriales ha permitido el intercambio de rasgos adaptativos entre las poblaciones. Sin embargo, este flujo génico puede complicar los esfuerzos por controlar Brettanomyces en la vinificación, donde su capacidad de resistencia a SO₂ y la producción de compuestos fenólicos volátiles es indeseable.
Impacto fenotípico de la diversidad genética
La diversidad genética de Brettanomyces se refleja no solo en su genoma sino también en sus rasgos fenotípicos, que tienen un impacto significativo en los procesos industriales. Uno de los rasgos fenotípicos más importantes es la producción de fenoles volátiles, como el 4-etilfenol y el 4-etilguaiacol, que son responsables del característico aroma a “Brett” en vino y cerveza (Suárez et al., 2007). Diferentes linajes genéticos de Brettanomyces varían en su capacidad de producir estos compuestos, lo que tiene consecuencias diferentes según el contexto productivo. En la elaboración de cerveza, especialmente en estilos tradicionales belgas como las cervezas Lambics y Gueuzes, estos compuestos son deseados ya que contribuyen a su característico sabor y aportan complejidad a su perfil aromático (Steensels et al., 2015). En contraste, en la vinificación, niveles elevados de estos compuestos son considerados un defecto que afecta negativamente a la calidad del vino (Renouf & Lonvaud-Funel, 2007).
Otro rasgo fenotípico de B. bruxellensis es la capacidad de formar biopelículas y de bioadhesión (adhesión a superficies). La capacidad de esta levadura para persistir en bodegas durante varios años, con contaminaciones recurrentes, sugiere que tiene propiedades específicas que le permiten sobrevivir en el entorno a través de fenómenos de bioadhesión. En un estudio realizado con más de 50 cepas representativas de los diferentes nichos de aislamiento de Brettanomyces, mostró que la mayoría de las cepas tenían una carga superficial negativa y eran hidrofílicas, mientras que un subgrupo de cepas de cerveza aisladas de vino exhibió un comportamiento hidrofóbico (Le Montagner et al., 2023). Por otro lado, también se estudió el efecto de factores bióticos y abióticos sobre la bioadhesión de Brettanomyces y se observó que la concentración de etanol y el pH parecía no tener un efecto significativo en las propiedades superficiales de la levadura, la formación de pseudohifas o la bioadhesión, mientras que los factores relacionados con la cepa y el grupo genético influenciaban fuertemente dichas propiedades (Le Montagner et al., 2024).
Resistencia al dióxido de azufre (SO₂)
La resistencia al SO₂, un conservante comúnmente utilizado en la elaboración de vino, es uno de los rasgos fenotípicos más estudiados en Brettanomyces (Figura 2). Se ha descrito que las cepas de Brettanomyces aisladas de entornos vinícolas tienen una mayor resistencia al dióxido de azufre (SO₂) que las cepas aisladas de cerveza o bioetanol, lo que demuestra cómo la plasticidad genética puede influir en la capacidad de adaptación a las condiciones productivas (Avramova, Vallet-Courbin, et al., 2018). Por ejemplo, cepas como L0308 y AWRI1499 han mostrado tener altos niveles de resistencia, lo que representa un desafío importante para la industria vinícola (Cibrario et al., 2020).
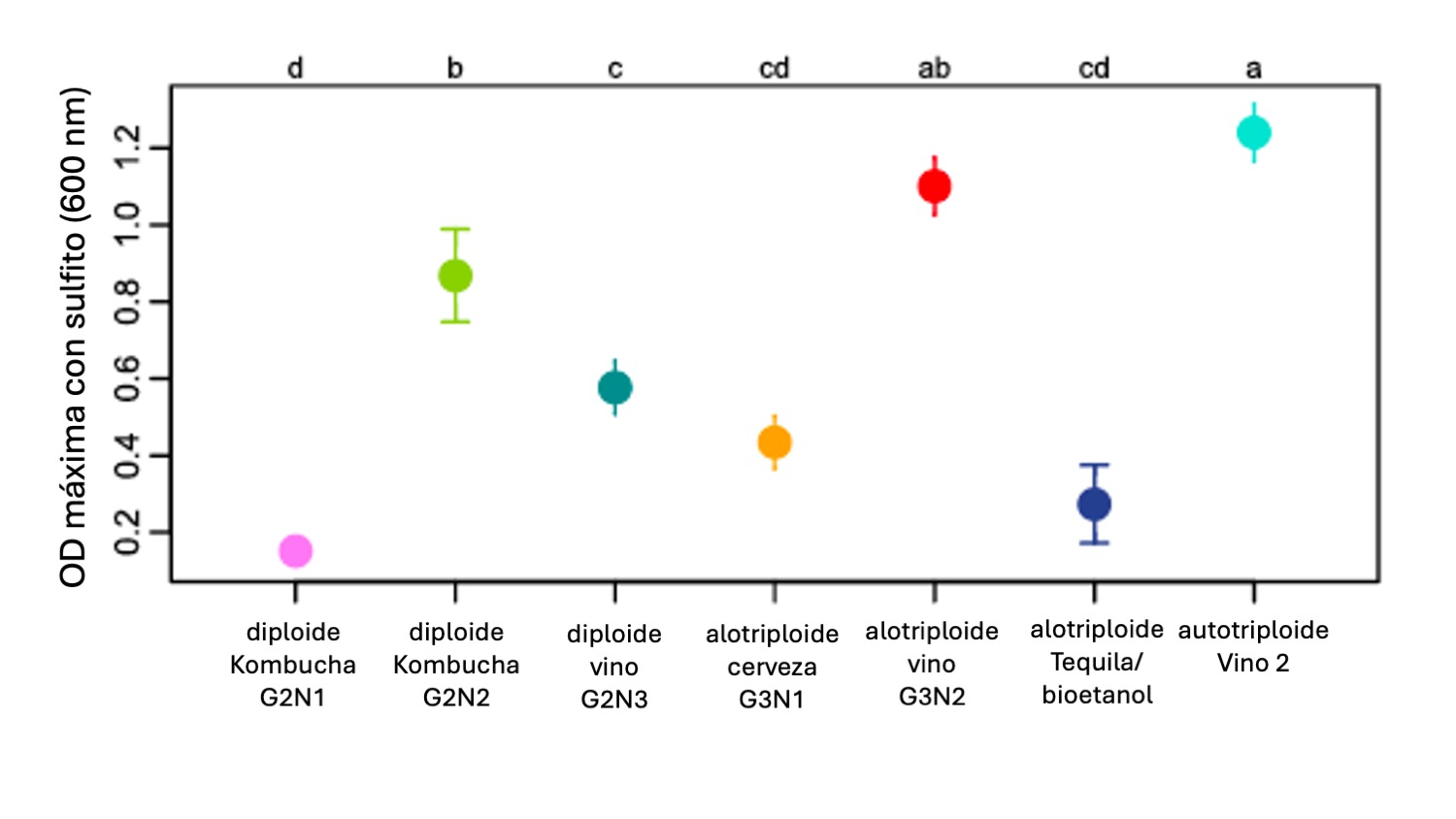
En otro estudio de Lleixà et al. (2021), 64 cepas de B. bruxellensis aisladas de vinos procedentes de barricas en Cataluña se evaluaron genética y fisiológicamente, y se observó una alta diversidad interespecífica que se correspondía con una gran variabilidad en la resistencia al SO₂ y en la producción de fenoles volátiles entre las cepas (Lleixà et al., 2021). De hecho, el estudio de Bartel et al. (2021) demostró la capacidad de B. bruxellensis para evolucionar adaptativamente en respuesta a la exposición a sulfito, con el gen SSU1 como principal mecanismo de resistencia, el cuál aumentaría en número de copias según la exposición a este compuesto.
Estudios recientes han comenzado a desentrañar la base genética de estas diferencias fenotípicas, pero está claro que la diversidad genética de Brettanomyces juega un papel crucial en su impacto en los resultados de la fermentación.
Conclusión
La diversidad genética de Brettanomyces, especialmente B. bruxellensis, es un factor clave en su capacidad de adaptación y supervivencia en una amplia gama de entornos productivos. Esta diversidad, impulsada por la variación en la ploidía, la plasticidad genómica y la hibridación, permite a la levadura prosperar en condiciones desafiantes, como fermentaciones de cerveza con alto contenido de etanol o vinos tratados con SO₂. Comprender la diversidad genética y fenotípica de Brettanomyces es esencial para gestionar su papel en la fermentación industrial, ya sea como un organismo de deterioro o como una herramienta valiosa para obtener perfiles de fermentación únicos. A medida que los investigadores desentrañen los fundamentos genéticos de la diversidad de Brettanomyces, se estará en mejor disposición para desarrollar estrategias que permitan utilizar o mitigar sus efectos, dependiendo del contexto productivo en el que se encuentre. El avance en el conocimiento de la biología evolutiva de esta levadura mediante el desarrollo de herramientas genéticas más sofisticadas permitirá optimizar su uso en procesos de fermentación y mitigar sus posibles desventajas.
Agradecimientos
El presente fue financiado por Ministerio de Ciencia, Innovación y Universidades de España (PID2022-137807OB-C21).
Bibliografía
Albertin, W., Panfili, A., Miot-Sertier, C., Goulielmakis, A., Delcamp, A., Salin, F., Lonvaud-Funel, A., Curtin, C., y Masneuf-Pomarede, I.: “Development of microsatellite markers for the rapid and reliable genotyping of Brettanomyces bruxellensis at strain level”. Food Microbiology; 2014, 42, 188–195. https://doi.org/10.1016/j.fm.2014.03.012
Antunovics, Z., Nguyen, H.-V., Gaillardin, C., y Sipiczki, M.: “Gradual genome stabilisation by progressive reduction of the Saccharomyces uvarum genome in an interspecific hybrid with Saccharomyces cerevisiae”. FEMS Yeast Research; 2005, 5(12), 1141–1150. https://doi.org/10.1016/j.femsyr.2005.04.008
Avramova, M., Cibrario, A., Peltier, E., Coton, M., Coton, E., Schacherer, J., Spano, G., Capozzi, V., Blaiotta, G., Salin, F., Dols-Lafargue, M., Grbin, P., Curtin, C., Albertin, W., y Masneuf-Pomarede, I.: “ Brettanomyces bruxellensis population survey reveals a diploid-triploid complex structured according to substrate of isolation and geographical distribution. Scientific”. Reports; 2018 8(1), 1–13. https://doi.org/10.1038/s41598-018-22580-7
Avramova, M., Vallet-Courbin, A., Maupeu, J., Masneuf-Pomarède, I., y Albertin, W.: “Molecular diagnosis of Brettanomyces bruxellensis’ sulfur dioxide sensitivity through genotype specific method”. Frontiers in Microbiology; 2018, 9, 1–9. https://doi.org/10.3389/fmicb.2018.01260
Bartel, C., Roach, M., Onetto, C., Curtin, C., Varela, C., y Borneman, A.: “Adaptive evolution of sulfite tolerance in Brettanomyces bruxellensis”. FEMS Yeast Research; 2018, 21(5). https://doi.org/10.1093/femsyr/foab036
Borneman, A. R., Zeppel, R., Chambers, P. J., y Curtin, C. D.: “Insights into the Dekkera bruxellensis Genomic Landscape: Comparative Genomics Reveals Variations in Ploidy and Nutrient Utilisation Potential amongst Wine Isolates”. PLoS Genetics; 2014, 10(2). https://doi.org/10.1371/journal.pgen.1004161
Cibrario, A., Miot-Sertier, C., Paulin, M., Bullier, B., Riquier, L., Perello, M. C., de Revel, G., Albertin, W., Masneuf-Pomarède, I., Ballestra, P., y Dols-Lafargue, M.: “ Brettanomyces bruxellensis phenotypic diversity, tolerance to wine stress and wine spoilage ability”. Food Microbiology; 2020, 87. https://doi.org/10.1016/j.fm.2019.103379
Claussen, N.H.: “ Eine Methode zur Anwendung von Hansens Reinzuchtsystem bei der Herstellung von englischen gelagerten Biersorten”. Wochenschr. Brau; 1904, 1904, 370–383.
Curtin, C. D., Bellon, J. R., Henschke, P. A., Godden, P. W., y De Barros Lopes, M. A.: “ Genetic diversity of Dekkera bruxellensis yeasts isolated from Australian wineries”. FEMS Yeast Research, 2007, 7(3), 471–481. https://doi.org/10.1111/j.1567-1364.2006.00183.x
Curtin, C., Kennedy, E., y Henschke, P. A.: “ Genotype-dependent sulphite tolerance of Australian Dekkera (Brettanomyces) bruxellensis wine isolates”. Letters in Applied Microbiology, 2012, 55(1), 56–61. https://doi.org/10.1111/j.1472-765X.2012.03257.x
Custers, M.T.J.: Onderzoekingen over het gistgeslacht Brettanomyces. 1940, DelftUniversity, Delft.
Eberlein, C., Saada, O. A., Friedrich, A., Albertin, W., y Schacherer, J.: “Different trajectories of polyploidization shape the genomic landscape of the Brettanomyces bruxellensis yeast species”. Genome Research, 2021, 31(12), 2316–2326. https://doi.org/10.1101/gr.275380.121
Fournier, T., Gounot, J. S., Freel, K., Cruaud, C., Lemainque, A., Aury, J. M., Wincker, P., Schacherer, J., y Friedrich, A.: “ High-quality de novo genome assembly of the Dekkera bruxellensis yeast using Nanopore MinION sequencing”. G3: Genes, Genomes, Genetics, 2017, 7(10), 3243–3250. https://doi.org/10.1534/g3.117.300128
Gallone, B., Mertens, S., Gordon, J. L., Maere, S., Verstrepen, K. J., y Steensels, J.: “ Origins, evolution, domestication and diversity of Saccharomyces beer yeasts”. Current Opinion in Biotechnology, 2018, 49, 148–155. https://doi.org/10.1016/j.copbio.2017.08.005
Goddard, M. R.: “Quantifying the complexities of Saccharomyces cerevisiae’s ecosystem engineering via fermentation”. Ecology, 2008 89(8), 2077–2082. https://doi.org/10.1890/07-2060.1
Gounot, J. S., Neuvéglise, C., Freel, K. C., Devillers, H., Piškur, J., Friedrich, A., y Schacherer, J.: “High complexity and degree of genetic variation in Brettanomyces bruxellensis population”. Genome Biology and Evolution, 2020, 12(6), 795–807. https://doi.org/10.1093/GBE/EVAA077
Harrouard, J., Eberlein, C., Ballestra, P., Dols-Lafargue, M., Masneuf-Pomarede, I., Miot-Sertier, C., Schacherer, J., y Albertin, W.: “ Brettanomyces bruxellensis: Overview of the genetic and phenotypic diversity of an anthropized yeast”. Molecular Ecology, 2023, 32(10), 2374–2395. https://doi.org/10.1111/mec.16439
Hellborg, L., y Piškur, J.:”Complex nature of the genome in a wine spoilage yeast, Dekkera bruxellensis”. Eukaryotic Cell, 2009, 8(11), 1739–1749. https://doi.org/10.1128/EC.00115-09
Le Montagner, P., Bakhtiar, Y., Miot-Sertier, C., Guilbaud, M., Albertin, W., Moine, V., Dols-Lafargue, M., y Masneuf-Pomar Ede, I.: “Effect of abiotic and biotic factors on Brettanomyces bruxellensis bioadhesion properties”. Food Microbiology, 2024, 120, 104480. https://doi.org/10.1016/j.fm.2024.104480
Le Montagner, P., Guilbaud, M., Miot-Sertier, C., Brocard, L., Albertin, W., Ballestra, P., Dols-Lafargue, M., Renouf, V., Moine, V., Bellon-Fontaine, M. N., y Masneuf-Pomarède, I.: “High intraspecific variation of the cell surface physico-chemical and bioadhesion properties in Brettanomyces bruxellensis”. Food Microbiology, 2023, 112. https://doi.org/10.1016/j.fm.2023.104217
Lebleux, M., Denimal, E., de Oliveira, D., Marin, A., Desroche, N., Alexandre, H., Weidmann, S., y Rousseaux, S.: “Prediction of genetic groups within Brettanomyces bruxellensis through cell morphology using a deep learning tool”. Journal of Fungi, 2021, 7(8). https://doi.org/10.3390/jof7080581
Libkind, D., Hittinger, C. T., Valeŕio, E., Gonca̧lves, C., Dover, J., Johnston, M., Gonca̧lves, P., y Sampaio, J. P.: “ Microbe domestication and the identification of the wild genetic stock of lager-brewing yeast”. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(35), 14539–14544. https://doi.org/10.1073/pnas.1105430108
Lleixà, J., Martínez-Safont, M., Masneuf-Pomarede, I., Magani, M., Albertin, W., Mas, A., y Portillo, M. C.: “Genetic and phenotypic diversity of Brettanomyces bruxellensis isolates from ageing wines”. Food Bioscience, 2021, 40. https://doi.org/10.1016/j.fbio.2021.100900
Oro, L., Canonico, L., Marinelli, V., Ciani, M., y Comitini, F.: “Occurrence of Brettanomyces bruxellensis on Grape Berries and in Related Winemaking Cellar”. Frontiers in Microbiology, 2019, 10(MAR). https://doi.org/10.3389/fmicb.2019.00415
Peynaud, E., y Domercq, S.: “Sur les Brettanomyces isolés de raisins et de vins”. Archiv fuer Mikrobiologie. 1956, 24, 266–280.
Piškur, J., Ling, Z., Marcet-Houben, M., Ishchuk, O. P., Aerts, A., LaButti, K., Copeland, A., Lindquist, E., Barry, K., Compagno, C., Bisson, L., Grigoriev, I. V., Gabaldón, T., y Phister, T.: “The genome of wine yeast Dekkera bruxellensis provides a tool to explore its food-related properties”. International Journal of Food Microbiology, 2012, 157(2), 202–209. https://doi.org/10.1016/j.ijfoodmicro.2012.05.008
Reis, A. L. S., de Souza, R. F. R., Torres, R. R. N. B., Leite, F. C. B., Paiva, P. M. G., Vidal, E. E., y de Morais Jr., M. A.: “Oxygen-limited cellobiose fermentation and the characterization of the cellobiase of an industrial Dekkera/Brettanomyces bruxellensis strain”. SpringerPlus, 2014, 3(1), 1–9. https://doi.org/10.1186/2193-1801-3-38
Renouf, V., y Lonvaud-Funel, A.: “Development of an enrichment medium to detect Dekkera/Brettanomyces bruxellensis, a spoilage wine yeast, on the surface of grape berries”. Microbiological Research, 2007, 162(2), 154–167. https://doi.org/10.1016/j.micres.2006.02.006
Roach, M. J., y Borneman, A. R.: “New genome assemblies reveal patterns of domestication and adaptation across Brettanomyces (Dekkera) species”. BMC Genomics, 2020, 21(1). https://doi.org/10.1186/s12864-020-6595-z
Schifferdecker, A. J., Dashko, S., Ishchuk, O. P., y Piškur, J.: “The wine and beer yeast Dekkera bruxellensis”. Yeast, 2014, 31(9), 323–332. https://doi.org/10.1002/yea.3023
Serpaggi, V., Remize, F., Recorbet, G., Gaudot-Dumas, E., Sequeira-Le Grand, A., y Alexandre, H.: “Characterization of the “ viable but nonculturable” (VBNC) state in the wine spoilage yeast Brettanomyces”. Food Microbiology, 2012, 30(2), 438–447. https://doi.org/10.1016/j.fm.2011.12.020
Steensels, J., Daenen, L., Malcorps, P., Derdelinckx, G., Verachtert, H., y Verstrepen, K. J.: “Brettanomyces yeasts – From spoilage organisms to valuable contributors to industrial fermentations”. International Journal of Food Microbiology 2015, 206, 24–38. https://doi.org/10.1016/j.ijfoodmicro.2015.04.005
Suárez, R., Suárez-Lepe, J. A., Morata, A., y Calderón, F.: “The production of ethylphenols in wine by yeasts of the genera Brettanomyces and Dekkera: A review”. Food Chemistry, 2007, 102(1), 10–21. https://doi.org/10.1016/j.foodchem.2006.03.030
VanderWalt, J.P.: Dekkera. In: Kreger-van Rij, N.J.W. (Ed.), The Yeasts: A Taxonomic Study, 3th edition Elsevier Science, Amsterdam, 1984. pp. 146–150.
Woolfit, M., Rozpedowska, E., Piskur, J. y Wolfe, K.H.: “Genome survey sequencing of the wine spoilage yeast Dekkera (Brettanomyces) bruxellensis”. Eukaryotic Cell 2007, 6, 721–733. https://doi.org/10.1128/ec.00338-06






