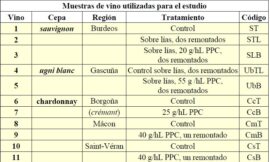
Oenococcus oeni (sin. Leuconostoc oenos) es una bacteria grampositiva perteneciente al grupo de las bacterias lácticas, que posee un cromosoma con un tamaño de 1932 kb para la cepa GM, según se ha deducido del análisis de fragmentos de macrorrestricción con diversos enzimas. La cepa
PSU-1, aislada originalmente en la Penn State University y usada comercialmente para iniciar la fermentación maloláctica, posee un tamaño de 1857 kb –según macrorrestricción–, o de 1820 kb –según la secuencia completa de esta cepa–. Ambas cepas representan dos grupos genómicos divergentes de acuerdo con los análisis de macrorrestricción (construcción de mapas que indican el orden entre los lugares en que enzimas de restricción cortan los cromosomas) y ribotipificación (método de biología molecular que se emplea con fines taxonómicos para obtener patrones genotípicos de las cepas, basándose en el análisis de su RNA ribosómico, altamente conservado durante la evolución). El contenido medio en GC (la evolución en el contenido en guanina + citosina es un indicador de la evolución de las proteínas y, por tanto, un parámetro filogenético) de la cepa PSU-1 es del 37,5 %.
En la actualidad se está abordando la secuenciación del genoma completo de O. oeni, proyecto en el que participan varios laboratorios de investigación franceses y americanos. Por el momento, se han obtenido datos completos de la cepa de O. oeni PSU-1, aunque aún se encuentran en formato de borrador. Disponer de tal información es importante, pero ahora hay que procesar todos esos datos y correlacionarlos, tanto con actividades metabólicas como con propiedades tecnológicas. Además, la secuencia del genoma de una cepa no va a proporcionar las respuestas a todas las preguntas que nos podemos plantear para todas las cepas y condiciones posibles. En cuanto a la identificación y caracterización de genes concretos, se han publicado las secuencias de varios genes de O. oeni, entre los que se encuentran los genes ribosómicos de las subunidades 16S y 23S, los genes del enzima maloláctico (mleA) y del transporte de ácido málico (mleP), los genes de la acetolactato sintetasa (alsS) y de la acetolactato descarboxilasa (alsD), genes de la ruta de utilización de la arginina, como la arginina desiminasa (arcA), ornitina transcarbamilasa (arcB) y carbamato quinasa (arcC), los genes de la histidina descarboxilasa (hdcA), los de varias proteínas de estrés como hps18, clpX, tras, genes que codifican para RNA de transferencia, como el gen del Ala-t-RNA, del Leu-t-RNA, genes que codifican para la síntesis del Leu-t-RNA, del operón de una permeasa de un oligopéptido y de un gen de la biosíntesis de un polisacárido y de la subunidad b’ de la DNA polimerasa (rpoC). Asimismo, se ha publicado la existencia de una secuencia de inserción IS1165, de genes relacionados con lugares de integración de fagos lisogénicos en el cromosoma bacteriano attB y de diversos fragmentos aleatorios obtenidos por PCR. Varios de estos genes se han localizado en los mapas genéticos de las cepas GM y PSU‑1.
Dificultad de manipulación genética
Con todo, no son demasiados los genes que están perfectamente identificados en esta especie, y dada su importancia biológica e industrial deberían existir más genes caracterizados, especialmente los relacionados con propiedades tecnológicas.
Debido a la dificultad de manipulación genética que presenta este organismo, hay pocos estudios que empleen este tipo de técnicas en O. oeni. Aunque se han encontrado algunos plásmidos en esta especie, en general no son frecuentes y la mayoría de ellos son crípticos. Por tanto, no parece que tengan implicaciones de importancia en propiedades tecnológicas de las cepas que los portan. También presentan escasas relaciones entre ellos, a excepción de unos pocos que, en realidad, podrían considerarse casi el mismo plásmido aislado varias veces por distintos autores. Su contenido en GC es cercano al de la bacteria, presentando mecanismos de replicación tipo círculo rodante unos de ellos, y bidireccional otros. Son plásmidos de características bastante particulares, incluso difíciles de clonar y estudiar en plásmidos convencionales de E. coli. Con todo, no se ha avanzado tampoco mucho en el conocimiento, y menos en la aplicación de estos plásmidos, o derivados de los mismos, a la genética de O. oeni. La existencia de plásmidos propios en esta especie debería permitir abordar, en principio, la construcción de vectores de clonación que se repliquen y se expresen en O. oeni, aunque hasta el momento no existen noticias de que esto se haya conseguido.
Algo parecido ocurre con los bacteriófagos (virus bacterianos) que atacan esta especie. Se han estudiado y caracterizado algunos fagos específicos de O. oeni, aunque no se han llegado a desarrollar todavía sistemas de manipulación genética basados en ellos; existen, sin embargo, algunos grupos que han trabajado o están trabajando para lograrlo. Es importante indicar que estos bacteriófagos presentan muchas características comunes entre sí, tanto morfológicas como genéticas o funcionales. Aunque un porcentaje importante de cepas de Oenococcus oeni son portadoras de fagos lisogénicos (un 70 %), y se han detectado fagos libres en vinos (20 % de los vinos), parece que los fagos de O. oeni ocasionan sólo excepcionalmente la lisis de las bacterias en vino. Por tanto, la mayoría de evoluciones irregulares de la fermentación maloláctica serían atribuibles a otras causas. De todas formas, ha sido posible obtener cepas de O. oeni resistentes a fagos, que mantienen tanto esa resistencia como características tecnológicas adecuadas durante muchas generaciones.
Plásmidos y transposones
Como hemos apuntado anteriormente, Oenococcus oeni presenta grandes dificultades de manipulación genética. Uno de los obstáculos más importantes para la consecución de este objetivo es que, hasta el momento, no se ha conseguido llevar a cabo la transformación genética de O. oeni. Los intentos han sido numerosos (y los grupos de investigación que se lo han propuesto también), pero hasta la fecha no se ha logrado en esta especie. Una vez más parece ser un organismo con un comportamiento genético muy particular, como se manifiesta al poseer plásmidos propios y fagos difíciles de manipular.
No obstante, nuestro grupo de investigación ha logrado introducir plásmidos conjugativos en esta bacteria: el plásmido pVA797 ha sido transferido desde Streptococcus sanguis a O. oeni a través de Lactococcus lactis. Las eficiencias de este proceso son muy bajas, y se han obtenido transconjugantes en los que el plásmido original ha sufrido varias deleciones y reorganizaciones, dando lugar a otro plásmido más pequeño denominado pLO1. Una mayor eficiencia de transconjugantes se consiguió al transferir el plásmido pIP501 desde S. sanguis a O. oeni vía L. lactis. Al igual que en el caso anterior, el plásmido que presentaban los transconjugantes era de menor tamaño, habiendo sufrido deleciones extensas en todos los casos, que afectaban a las regiones de transferencia conjugativa y la estabilidad de los plásmidos originales. Como dato curioso del comportamiento de estos plásmidos reorganizados y delecionados inestables en O. oeni, hemos visto que al ser transferidos a L. lactis presentan una estabilidad completa en esta bacteria láctica. En otras palabras, ha sido posible introducir plásmidos conjugativos en O. oeni, pero con una baja eficiencia y dando como producto unos plásmidos menores y bastante inestables. En esta ocasión, volvemos a encontrarnos con particularidades de O. oeni que dificultan una manipulación genética sencilla y eficaz.
Nuestro grupo de investigación también ha logrado introducir por conjugación en O. oeni los transposones Tn916 y Tn925. Estos transposones se integran en distintos sitios del cromosoma de O. oeni y no sufren aparentemente transposiciones secundarias. En este caso, la estabilidad es elevada. Todo ello, junto con el conocimiento que vamos poseyendo de éstos y otros transposones, posibilitaría el desarrollo de vehículos de manipulación genética basados en los mismos. Por el momento, seguimos a la espera, y estos dos métodos conjugativos, mediante plásmidos y transposones, son los únicos sistemas disponibles de introducción de DNA exógeno en Oenococcus oeni descritos hasta la fecha.
Mutación maloláctica
En cuanto a la obtención de cepas mutantes mediante sistemas que no impliquen DNA recombinante, hemos podido aislar en nuestro equipo mutantes malolácticos que carecen por completo de esta actividad. Por un lado, estos mutantes permiten estudios fisiológicos de interés básico y tecnológico, y por otro permiten la utilización de sistemas aprobados para manipulación de organismos de interés alimentario como O. oeni. En contrapartida, también hemos desarrollado sistemas no recombinantes que nos permiten aislar cepas de O. oeni con capacidad de fermentación maloláctica aumentada.
Las levaduras crecen con mucha facilidad en el mosto en fermentación, mientras que las bacterias lo hacen con mucha dificultad y mucho más lentamente. Es por ello que la posibilidad de tener una levadura capaz de hacer la fermentación maloláctica siempre ha llamado la atención. Una vez desarrolladas las herramientas adecuadas para levaduras, los primeros intentos de construir una levadura maloláctica mediante técnicas de ingeniería genética fueron en 1984, al clonar el gen maloláctico de L. delbrueckii en Saccharomyces cerevisiae. La actividad detectada en la cepa transformada fue muy baja, ya que tan sólo un 1 % de L-malato se transformaba en L-lactato. Posteriormente se construyó una genoteca de O. oeni en una cepa de E. coli incapaz de usar L-malato. Los clones recombinantes fueron inestables y no se llegó a averiguar si eran capaces o no de llevar a cabo la fermentación maloláctica. En 1993 y 1994 se logró clonar el gen maloláctico de Lactococcus lactis en E. coli y en S. cerevisiae. En ambos casos se demostró que las cepas de E. coli y de S. cerevisiae transformaban ácido L-málico en ácido L-láctico, pero con una muy baja eficacia. Posteriormente, en 1997, se construyó una levadura maloláctica recombinante que coexpresaba el gen de la malato permeasa de Schizosaccaromyces pombe y el gen maloláctico de Lactococcus lactis. A diferencia de las levaduras malolácticas previamente desarrolladas, ésta era capaz de completar la fermentación maloláctica en menos de siete días en mostos de la variedad chardonnay. En este caso, la clonación de la malato permeasa permitió que el transporte del ácido málico al interior de la levadura dejase de ser el paso limitante en el proceso de degradación del ácido málico en la levadura maloláctica. Con todo, el haber logrado que una levadura realice la fermentación maloláctica no supone la eliminación de las bacterias lácticas para realizar este proceso en bodega. Además del hecho que las bacterias lácticas proporcionan otros metabolitos de importancia organoléptica, aparte de la conversión del ácido málico a ácido láctico, también le confieren una mayor estabilidad microbiológica a los vinos en los que se desarrollan. Por otro lado, si una levadura efectúa la fermentación maloláctica al mismo tiempo que la alcohólica, ambos procesos no pueden desacoplarse, ni interrumpirse de forma independiente, y ello significa que o bien el ácido málico se degrada siempre completamente, o se interrumpe la fermentación alcohólica.
Sistemas de tipificación
Finalmente, en cuanto a los sistemas moleculares que se han empleado en la especie Oenococcus oeni, merece la pena indicar el desarrollo de sistemas de identificación o de tipificación para esta especie, y un buen ejemplo lo tenemos en un artículo de A. Bordons et al. en este mismo número de la revista. Muchas técnicas se han desarrollado para ello, entre las que indicamos los RAPD (amplificación aleatoria de DNA polimórfico), RFLP-PFGE (polimorfismo en la longitud de los fragmentos de amplificación analizado por electroforesis en campo pulsante), 16S-ARDRA (análisis de restricción del DNA ribosómico 16S amplificado), FISH (hibridación in situ con fluorescencia), etc. La mayoría de ellas parecen indicar que la diversidad intraespecífica de O. oeni es baja, aunque hay indicios que podrían sugerir la existencia de dos subespecies. También se ha intentado relacionar datos de técnicas moleculares con propiedades tecnológicas u orígenes geográficos de las cepas aisladas. Por el momento, la información que han obtenido distintos grupos de investigación es no sólo parcial, sino incluso contradictoria, por lo que es necesario profundizar más y con herramientas mejores y más potentes que las empleadas hasta el momento.
En conclusión, queda por conocer lo más interesante en la genética de Oenococcus oeni. Pensemos que nos hallamos al inicio del camino, y hasta el momento no se han descrito apenas manipulaciones genéticas en esta especie. Cuando podamos realmente controlar la genética de este organismo, podremos plantear conocer mejor fisiológicamente esta bacteria e incidir en sus propiedades tecnológicas. Ello, sin duda, nos permitirá trabajar con cepas mejores, más resistentes, más seguras, más rápidas, y con propiedades mejoradas respecto a las actuales.
| GLOSARIO
bacteriófago: partícula de tipo vírico que infecta y lisa –destruye– bacterias, extendiéndose mediante la liberación de nuevas partículas víricas maduras de las células lisadas. deleción: supresión de una porción de un gen. lisogénico: uno de los dos tipos de ciclo de vida de un bacteriófago (lítico y lisogénico) por el que no produce partículas fágicas, llegando normalmente el DNA del fago a formar parte del cromosoma bacteriano en forma de profago. macrorrestricción: construcción de mapas que indican el orden entre los lugares en que enzimas de restricción cortan los cromosomas. plásmido conjugativo: plásmido que transfiere genes de una célula procariótica a otra por un mecanismo de contacto entre ellas. plásmido: elemento genético extracromosómico bacteriano, que no es esencial para el crecimiento de la célula y que no tiene forma extracelular. ribotipificación: método de biología molecular que se emplea con fines taxonómicos para obtener patrones genotípicos de las cepas, basándose en el análisis de su RNA ribosómico, altamente conservado durante la evolución. transformación: transferencia de información genética por medio de DNA libre. transposón: elemento genético que puede desplazarse de un lugar a otro en un cromosoma y que, además de transportar genes de la transposición, lleva algunos otros; a menudo, son genes que producen fenotipos que pueden ser seleccionados, por ejemplo, resistencia a antibióticos. |