La estructura genética de las variedades de vid españolas está relacionada con la larga y compleja historia de su cultivo, que se remonta a los pueblos celtas, íberos y fenicios. Durante todo este tiempo, el empleo de las variedades y clones más adaptados a las condiciones ambientales y a los gustos del consumidor de cada época ha contribuido a la distribución generalizada de genotipos, y a la confusión de nombres empleados para denominar a cada variedad en las diferentes regiones. La descripción morfológica de las variedades [ampelografía (IPGRI, UPOV, OIV, 1997)] ha permitido abordar la caracterización varietal y, de esta forma, discriminar plantas que, pese a denominarse de forma diferente, pertenecen a una misma variedad (sinonimias) y plantas que compartiendo nombre pertenecen a variedades distintas (homonimias). En la actualidad, múltiples herramientas moleculares, como isoenzimas, RFLP, RAPD y microsatélites, permiten diferenciar variedades (Karp y Edwards, 1998), y son ampliamente utilizadas para identificar y distinguir las variedades de vid.
Origen de la variabilidad intravarietal
Es bien conocido por agricultores y viveristas que existe heterogeneidad, tanto en el comportamiento agronómico como en rasgos morfológicos, entre clones pertenecientes a una misma variedad. Se ha postulado que esta variabilidad puede tener tres orígenes (Martínez de Toda, 1991):
Origen policlonal: Los clones que constituyen una variedad proceden de semillas diferentes, aunque mantienen caracteres comunes, responsables de que sean reconocidos como pertenecientes a la misma variedad. Estas variedades se han denominado con el término de variedad-población.
- Origen monoclonal: Los diferentes clones de una variedad derivan de una única semilla original y la variabilidad existente es debida a la acumulación de mutaciones somáticas.
- Origen patogénico: Las diferencias morfológicas son consecuencia de las alteraciones provocadas por la infección con algún patógeno, como por ejemplo una infección vírica.
Los resultados obtenidos de los estudios de las relaciones genéticas entre variedades de vid muestran la existencia de elevados niveles de polimorfismo intervarietal. Por este motivo, cuando el origen de una variedad es policlonal, los distintos genotipos que integran la variedad deberían distinguirse fácilmente con cualquier marcador molecular. Sin embargo, los clones pertenecientes a una variedad muestran un patrón de bandas común para la mayoría de marcadores utilizados, como isoenzimas, RFLP, microsatélites, RAPD e SIR, entre otros (Bowers et al., 1993; Botta et al., 1995, Loureiro et al., 1998; Moreno et al., 1998), resultados que apoyan el origen monoclonal de la mayor parte de las variedades. Por lo tanto, el origen policlonal de una variedad probablemente corresponde a variedades pobremente caracterizadas genética y morfológicamente y que, como consecuencia, incluyen distintas homonimias Si, como parece razonable pensar en base a los resultados acumulados, el origen de la mayor parte de las variedades es monofilético, es decir, si cada variedad deriva de una única semilla, los clones propagados vegetativamente a partir de la planta original van acumulando cambios en su secuencia genética (mutaciones somáticas) que, si afectan a caracteres de interés agronómico, pueden dar lugar a una nueva variedad. Ejemplos que ilustran esta situación son las variedades pinot noir, pinot gris y pinot blanc (Regner et al., 2000), o las variedades garnacha, garnacha gris o dorada, garnacha blanca (J.A. Cabezas et al., resultados sin publicar), que desde un punto de vista genético son indistinguibles si se emplean isoenzimas, microsatélites y RAPD.
Análisis de la diversidad intravarietal basado en el empleo de marcadores de alta eficacia
Las técnicas de marcadores empleadas para discriminar variedades no son las más adecuadas para diferenciar las diferentes variantes somáticas que se acumulan en cada variedad y que son el origen de los clones, entendiendo como clon las plantas obtenidas a partir de una misma cepa madre (Reyner, 1991). Estos marcadores sólo permiten analizar un número reducido de loci por genoma, lo que no es suficiente para detectar diferencias entre clones pertenecientes a una misma variedad aunque, algunos estudios basados en RAPD y microsatélites indicaban la existencia de polimorfismos intravarietales (Silvestroni et al., 1997; Regner et al., 2000). Sin embargo, el reciente desarrollo de marcadores de alta eficacia permite abordar el estudio de la variabilidad intravarietal, gracias a la posibilidad de analizar miles de secuencias de fragmentos anónimas por genoma. Entre estos marcadores, los AFLP, desarrollados en 1995 por Vos et al., y marcadores derivados, como SAMPL y S-SAP (Paglia y Morgante, 1998; Waugh et al., 1997), pueden ser de utilidad para identificar polimorfismos entre clones pertenecientes a diferentes variedades.
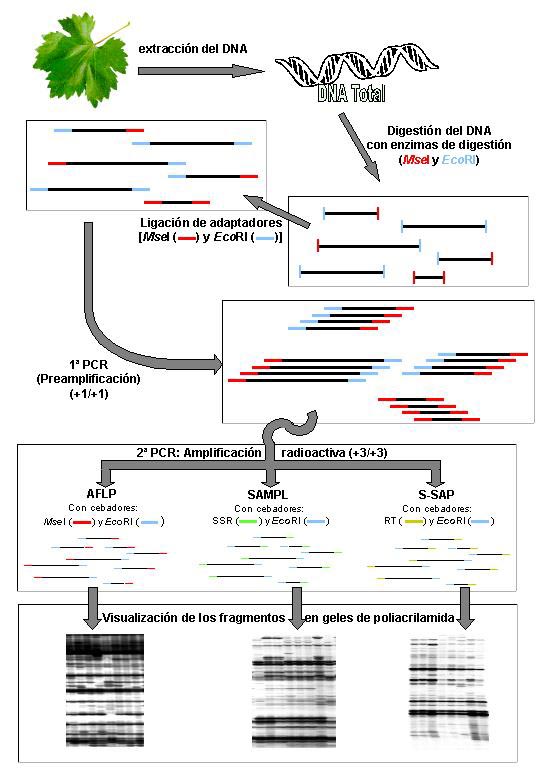
La técnica AFLP se basa en la amplificación selectiva de fragmentos de DNA obtenidos mediante digestión con enzimas de restricción (Vos et al., 1995). Combina, por lo tanto, la especificidad proporcionada por la digestión con enzimas, que reconocen secuencias específicas del genoma, con la rapidez y facilidad de detección de polimorfismos mediante amplificación PCR. Modificando los nucleótidos de selección se amplifican grupos de fragmentos diferentes, por lo que el empleo de la técnica AFLP permite la identificación de un número ilimitado de marcadores. La amplificación AFLP es específica y reproducible al usar cebadores complementarios a la secuencia de los adaptadores que se ligan a los extremos de los fragmentos de DNA digeridos. La presencia o ausencia de cada banda obtenida genera un código de barras o patrón que es específico para cada muestra analizada. Con objeto de aumentar el nivel de polimorfismo genético se han desarrollado marcadores moleculares basados en la tecnología AFLP pero que amplifican regiones del genoma que contienen microsatélites, mediante el empleo en la amplificación selectiva de un cebador AFLP y un cebador microsatélite con anclaje 5’ o 3’ (marcadores SAMPL, del inglés Selective Amplification of Microsatellite Polymorphic Loci), o regiones que contienen retrotransposones, mediante el empleo de un cebador AFLP y un cebador basado en una secuencia conservada de los retrotransposones (marcadores S-SAP, de Sequence-Specific Amplification Polymorphisms). En la figura 1 se muestra un esquema de las tres técnicas de marcadores de alta eficacia (AFLP, SAMPL y S-SAP) y un detalle de los perfiles de AFLP, SAMPL y S-SAP obtenidos para diferentes muestras de vid.
El empleo de marcadores de alta eficacia permite la diferenciación e identificación rápida de variedades, incluso cuando se trata de variedades estrechamente relacionadas entre sí. Además, estos marcadores permiten también detectar diferencias entre clones pertenecientes a la misma variedad (Ribaut et al., 1997; Cervera et al., 1998; Scott et al., 2000). Empleando AFLP (figura 2) y SAMPL, se detectaron diferencias entre clones pertenecientes a las variedades Flame y Napoleón (Cervera et al., 2000b y resultados sin publicar), o entre clones de variedades conservadas en diferentes colecciones de vid, como por ejemplo: albillo, turruntés, malvasía del Bierzo, tempranillo, garnacha, moscatel de grano menudo, moscatel de Málaga, albariño, airén, malvar, macabeo, parellada, xarel.lo (Cervera et al., 2001; Cervera et al., enviado; y resultados sin publicar). El estudio de diversidad genética intravarietal reveló que variedades multiplicadas activamente en vivero, como por ejemplo tempranillo, presentan niveles de variabilidad mucho menores que variedades multiplicadas por los agricultores, como es el caso de albillo, turruntés o malvasía del Bierzo (Cervera et al., enviado). Este resultado indica que la modificación de la estructura del viñedo actual, iniciada con el ataque de filoxera, y debida hoy en día al empleo de prácticas de cultivo basadas en la utilización de material multiplicado y distribuido por viveros, está causado una creciente erosión genética. Esta erosión se traduce en la pérdida de variabilidad genética del viñedo y, por lo tanto, conlleva el riesgo de desaparición de genotipos autóctonos, es decir, tanto variedades minoritarias como clones pertenecientes a variedades mayoritarias pero cuyo cultivo se reduce a una mínima proporción de genotipos seleccionados.
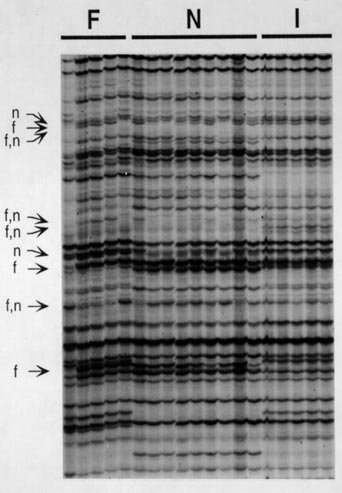
Con objeto de abordar el estudio del origen y de la estabilidad de la variación somática en la vid, estamos analizando material procedente de diferentes brazos de cepas centenarias pertenecientes a las variedades tempranillo, garnacha y albariño. Este estudio ha permitido identificar polimorfismos entre brazos de una misma cepa debidos a mutaciones acumuladas desde que se originaron a partir de meristemos vegetativos comunes, durante más de 100 años (Cervera et al., 2000a, y resultados sin publicar). En cada variedad podían detectarse dos clases de polimorfismos: fragmentos identificados en ramas de distintas cepas (polimorfismos recurrentes) y fragmentos presentes o ausentes en una única rama de una de las cepas (polimorfismos sencillos). Los AFLP revelaban un menor número de fragmentos polimórficos que los SAMPL y los S-SAP. El análisis molecular del origen de estos polimorfismos y de su distribución en las cepas de la variedad permite estudiar el origen de la variación somática y su estabilidad. Por otra parte, la comparación de los resultados obtenidos en las distintas variedades permite conocer si la de la tasa de la variación somática es diferente para cada variedad.
Aplicación de polimorfismos intravarietales en la mejora de variedades de vinificación
El cultivo y mejora de la uva de vinificación está reglamentado por los consejos reguladores de las denominaciones de origen, siendo la selección clonal la única estrategia de mejora de dichas variedades que se aborda en países de larga tradición vitivinícola como España, Francia, Italia, o Portugal. El análisis de la variabilidad genética existente entre distintas cepas de una variedad puede generar información de interés para el diseño de programas de selección clonal, al permitir identificar y seleccionar los genotipos más divergentes, que podrían emplearse como plantas madres, con objeto de incrementar la diversidad genética de partida.En caso de disponer de marcadores ligados a las regiones del genoma que controlan caracteres de interés (como por ejemplo los relacionados con síntesis de antocianos, terpenos, resveratrol) se podrían seleccionar los genotipos con el o los caracteres deseados y concentrar sobre ellos el trabajo de selección clonal. Finalmente, si se demuestra la especificidad y la estabilidad de algunos marcadores presentes en variantes somáticas (clones) de interés, estos marcadores podrían utilizarse para la identificación molecular de dichos clones. Sin embargo, todavía se requiere más información sobre estos polimorfismos intravarietales para que puedan ser de utilidad en la identificación y certificación clonal.
Agradecimientos
Este trabajo comenta algunos de los resultados de la caracterización de colecciones españolas de variedades de vid mediante AFLP, SAMPL y S-SAPs realizada en colaboración con los siguientes investigadores y grupos de investigación: Félix Cabello, de la Sección de Viticultura y Enología, IMIA (Alcalá de Henares, Madrid); José Luis Cénis y Juan Carreño, del CIDA (Murcia); Juan Carlos Sancha y Fernando Martínez de Toda, de la Universidad de La Rioja (Logroño, La Rioja); Teodoro Vicente, Juana Martínez y Teresa Martínez, del CIA (La Rioja); Carmen Martínez, José Ramón Vidal, de la Misión Biológica de Galicia (Pontevedra).
Nuestra investigación en vid está financiada por el proyecto 07B-0033-1999 de la Comunidad Autónoma de Madrid y por el Proyecto PETRI 0282-CT.
Bibliografía
Botta, R., Scott, N.S., Eynard, I., Thomas, M.R.:. «Evaluation of microsatellite sequence-tagged site markers for characterizing Vitis vinifera cultivars», Vitis 1995, 2: 99-102.
Bowers, J.E., Bandman, E.B., Meredith, C.P.: «DNA fingerprint characterization of some wine grape cultivars», Am J Enol Vitic 1993, 44: 266-274.
Cervera, M.T., Cabezas, J.A., Sancha, J.C., Martínez de Toda, F., Martínez-Zapater, J.M.: «Application of AFLP to the characterization of grapevine Vitis vinifera L. Genetic resources. A case study with accessions from La Rioja (Spain)», Theor Appl Gent 1998, 97: 51-59.
Cervera, M.T., Cabezas, J.A., Cenis, J.L., Zapater, J.M.: «Somatic variation in grapevine», Plant & Animal Genome Conference VIII 2000a, San Diego, California, Estados Unidos.
Cervera, M.T., Cabezas, J.A,. Sánchez-Escribano, E., Cenis, J.L., Martínez-Zapater, J.M.: «Genetic identification of table grape cultivars (Vitis vinifera L.) based on AFLP markers», Vitis 2000b, 39: 109-114.
Cervera, M.T., Rodrigez, I., Cabezas, J.A., Chávez, J., Martínez-Zapater, J.M., Cabello, F.: «Morphological and molecular characterization of grapevine accessions known as Albillo», Am J Enol Viticult 2001, 52 (en prensa).
Cervera, M.T., Rodrigez, I., Cabezas, J.A., Chávez, J., Cabello, F., Martínez-Zapater, J.M.: «Varietal diversity within grapevine accessions known as Tempranillo, a representative Spanish red wine variety» (enviado).
IPGRI, UPOV, OIV: «Descriptors for grapevine (Vitis spp.)», International Union for te Protection of New Varieties of Plants, Geneva, Switzerland / Office International de la Vigne et du Vin, Paris, France / International Plant Genetic Resources Institute, Rome, Italy, 1997.
Karp, A., Edwards, K.J.: «DNA markers: a global overview», en: DNA markers: protocols, aplications and overviews. Eds: G. Caetano- Anollés y P.M. Gresshoff, Wiley, Nueva York, 1998: 1-13.
Loureiro, M.D., Martínez, M.C., Boursiquot, J.M., This, P.: «Molecular marker analysis of Vitis vinifera ‘Albariño’ and some similar grapevine cultivars», J Amer Soc Hort Sci 1998, 132: 842-848.
Martínez de Toda, F.: «Biología de la vid. Fundamentos biológicos de la viticultura», Ediciones Mundi-Prensa, Madrid, 1991.
Moreno, S., Martín, J.P., Ortiz, J.M.: «Inter-simple sequence repeats PCR for characterization of closely related grapevine germplasm», Euphytica 1998, 101:117-125.
Paglia, G., Morgante, M.: «PCR-based multiplex DNA fingerprinting techniques for the analysis of conifer genomes», Mol Breed 1998, 4: 173-177.
Regner, F., Stadlbauer, A., Eisenheld, C., Kaserer, H.: «Genetic relationships among pinots and related cultivars», Am J Enol Viticult 2000, 51: 7-14.
Reyner, A.: «Manuel de Viticulture», Lavoisier, París 1991.
Ribaut, J.M., Thome, J., This, P., Gaitan, E., Clot, B., Perotti, E., Kupfer, P.: «AFLP: A powerful tool to identify polymorphisms in grapevine among both cultivars and clones», Plant & Animal Genome Conference V 1997, San Diego, California, Estados Unidos.
Scott, K.D., Ablett, E.M., Lee, L.S., Henry, R.J.: «AFLP markers distinguishing an early mutant of Flame Seedless grape», Euphytica 2000, 113: 245-249.
Silvestroni, O., Di Pietro, D., Intrieri, C. Vignani, R., Filippetti, I., Del Casino, C., Scali, M., Cresti, M.: «Detection of genetic diversity among clones of cv. Fortana (Vitis vinifera L.) by microsatellite DNA polymorphism analysis», Vitis 1997, 36: 147-150.
- Vos, P., Hogers, R., Bleeker, M., Reijans, M., van de Lee, T., Hornes, M., Frijters, A., Pot, J., Peleman, J., Kuiper, M., Zabeau, M.: «AFLP: A new technique for DNA fingerprinting», Nucleic Acids Res 1995, 23:4407-4414.
Waugh, R., McKelan, K., Flavell, A.J., Pearce, S.R., Kumar, A., Thomas, B.B.T., Powell, W.: «Genetic distribution of Bare-1-like retrotransposable elements in the barley genome revealed by sequence-specific amplification polymorphisms (S-SAP)», Mol Gen Genet 1997, 253: 687-694.