Para cualquier científico, biotecnología es el uso de un organismo vivo o una de sus partes o componentes para producir algo que luego se comercializa. Cuando el organismo vivo se usa para producir un alimento o una bebida se realiza biotecnología de alimentos. Desde este punto de vista, el vino es un producto de la biotecnología alimentaria ya que su producción no es más que la fermentación por parte del hombre (Homo sapiens) de un mosto obtenido desde los frutos de la vid (Vitis vinifera) por parte de levaduras (Saccharomyces cerevisiae). Esta es la visión de los científicos, pero dista mucho de la que tienen los consumidores europeos que piensan que biotecnología de alimentos es aplicar genética en la producción de los alimentos. Aun aceptando esta asunción incorrecta del concepto biotecnología, vino, biotecnología y genética están unidas desde hace miles de años.
El vino y la biotecnología
Recordemos que los primeros testimonios arqueológicos del cultivo de la vid datan de 7000 años a.C. en la zona de Mesopotamia. Desde entonces hasta nuestros días se ha producido vino en muchas partes del planeta a partir de distintos cultivares del género Vitis. La mayoría de especies utilizadas por la viticultura actual pertenecen a la especie Vitis vinifera, que es la especie de uva con mayor extensión cultivada. La domesticación de esta planta tuvo lugar hace 5000 años. Las adaptaciones más importantes fueron el cambio a la bisexualidad funcional y el aumento de tamaño del fruto.1 Todos estos cambios fenotípicos se produjeron cuando los agricultores aplicaron empíricamente la selección natural de mutaciones arbitrarias. También se han llevado a cabo programas de mejora aplicando cruzamientos sexuales forzados con la intención de conseguir una mayor resistencia al ataque de plagas o a estreses abióticos. Con respecto a la levadura vínica también durante años también se ha empleado de forma empírica genética en su mejora. De hecho, mientras que las cepas de laboratorio de esta especie tienen 16 cromosomas y un tamaño de genoma en torno a las 12 Mb de DNA, las cepas vínicas industriales suelen ser diploides o aneuploides o, en ocasiones, poliploides. El origen genético de las cepas vínicas actuales es motivo de controversia entre muchos grupos de investigación. Lo que parece evidente es que el genoma actual de las levaduras vínicas es el resultado de una fuerte presión de selección durante millones de generaciones.2 Además, durante los últimos veinte años se han intentado llevar a cabo mejoras genéticas dirigidas en cepas vínicas mediante el empleo de mutagénesis y selección, hibridación, citoducción o fusión de protoplastos, aunque muchos de estos abordajes no han sido satisfactorios desde el punto de vista industrial.3 En 2004, en la DOC Priorat existían unas 85 bodegas que gestionaban entre 8000 y 200 000 kg de uva, siendo la producción total de aproximadamente 4 360 000 kg de uva (el equivalente a 29 000 hL de vino).
Por todo lo expuesto podemos concluir que los organismos que intervienen en la producción del vino han sufrido procesos de mejora genética. Esas modificaciones han sido introducidas mediante procedimientos clásicos de mejora genética. Desde hace unos años los mejoradores disponen de las técnicas de la ingeniería genética que permiten aislar genes concretos, modificarlos en el laboratorio y reintroducirlos en el organismo original, o en otro distinto, generando los llamados organismos transgénicos u organismos modificados genéticamente (abreviadamente OMG). La enología y la viticultura no han sido ajenas a estos desarrollos y se ha comenzado a aplicar ingeniería genética sobre vid y sobre levaduras vínicas, aunque con diferente esfuerzo y resultados.
Genómica e ingeniería genética de la uva
La base de la ingeniería genética es la capacidad de poder aislar genes desde un genoma y disponer de sistemas de transformación genética que permitan introducir el gen clonado en un organismo distinto o reintroducirlo en el original. Para ello se precisa disponer de un conocimiento genómico y de técnicas de transformación genética. En el caso de V. vinifera se han comenzado diversos proyectos de secuenciación genómica. Muy recientemente, el Consorcio Público Italo-Francés para la Caracterización del Genoma de la Uva ha hecho públicos parte de sus resultados (The French-Italian Public Consortium for Grapevine Genome Characterization).4 Aparentemente, la variedad pinot noir PN40024 contiene en su genoma 30 434 genes, 149 351 exones y 118 917 intrones. Otro consorcio internacional, el Grapevine Genome Initiative, ha publicado la secuenciación del genoma del clon pinot noir ENTAV 115.5 En esta caso predicen un tamaño de genoma de 504,6 Mb y un total de 29 585 genes. La conclusión de estos primeros datos genómicos es clara: se han detectado multitud de genes ligados a propiedades físico-químicas, organoléptica o nutricionales con los que poder hacer proyectos de mejora genética asistida por marcadores. Existe otro resultado sorprendente de estos trabajos ya que se puede deducir de los mismos que el genoma actual de la uva es el resultado de la interacción de tres genomas haploides ancestrales que formaron lo que técnicamente podemos llamar un genoma ancestral paleohexaploide. En otras palabras, un transgénico natural.
En cuanto a la transformación genética de la uva, en general, los distintos cultivares de V. vinifera son muy recalcitrantes a la toma de DNA, fundamentalmente por la aparición de necrosis durante el proceso de regeneración tisular. No obstante, existen diversos trabajos que demuestran la posibilidad de obtener transformantes con baja eficiencia usando el método de transformación mediado por Agrobacterium6 o el de biolística,7 siendo de destacar que algunas de estas transformaciones se han llevado a cabo utilizando cultivares de vid de gran interés comercial como cabernet sauvignon, chardonnay, chenin blanc, riesling, sauvignon blanc, shiraz o muscat gordo blanco. En resumen, actualmente, la aplicación de las técnicas de la ingeniería genética en la mejora genética de la vid se encuentra en sus primeros estadios y en el futuro será necesario perfeccionar las técnicas de transformación e incrementar el conocimiento básico de esta planta para definir estrategias moleculares de mejora por ingeniería genética.8 Para ello resultará de especial relevancia el desarrollo de futuros proyectos de genómica comparativa.
Genómica e ingeniería genética de la levadura vínica
Saccharomyces cerevisiae es la especie microbiana más importante en la transformación del mosto de uva en vino. Es la misma especie responsable de la producción del pan, la cerveza, los destilados alcohólicos y, fuera del ámbito occidental múltiples procesos fermentativos de productos naturales. Esta diversidad de aplicaciones es indicativa de la diversidad genética y fenotípica que puede albergar esta especie. De hecho durante años se ha explotado esta diversidad genética en el ámbito enológico dando lugar a la selección de cepas apropiadas para diversos estilos de vinificación, características fisicoquímicas de los mostos, o buscando la tipicidad de los vinos.
Por otra parte, S. cerevisiae ha sido y es un organismo modelo, cuyo estudio y manipulación están, en mayor o menor medida, en la base del desarrollo de muchas de las grandes disciplinas científicas dentro del ámbito de la biología. Muchos de los avances metodológicos recientes de la biotecnología, tales como la genómica, proteómica o metabolómica, también han utilizado S. cerevisiae como organismo modelo. Esta posición, junto con la ventaja adicional de ser el modelo más sencillo de célula eucariota, hace a su vez que se hayan desarrollado multitud de herramientas biotecnológicas que permiten el estudio y la manipulación de esta especie. La secuencia del primer genoma completo de una cepa de S. cerevisiae fue publicado hace más de 12 años,9 existen multitud de vectores desarrollados para su transformación genética, plataformas específicas para el análisis genómico y proteómico, y colecciones de cepas con genotipo totalmente caracterizado, que facilitan enormemente el análisis de la función de cada gen concreto. Mediante estudios genómicos se ha revelado por ejemplo algunas claves de la evolución reciente de las levaduras vínicas;10 incluyendo la adquisición de resistencia a sulfitos.11 Varios estudios transcriptómicos han permitido tener una mejor comprensión de cómo los diferentes factores de estrés afectan a la expresión génica durante la fermentación.12-17
A pesar de los intentos de algunos grupos de investigación, la explotación comercial de estas herramientas biotecnológicas en la fermentación alcohólica del mosto de uva, ha seguido una marcha parsimoniosa. Existen múltiples explicaciones para esta lentitud, que van desde la reticencia general de los consumidores a aceptar productos derivados de la biotecnología en su dieta, hasta las propias características del sector, que basa parte de su valor añadido en la imagen de producto tradicional. No en vano, los cambios en esta tendencia se están desarrollando fundamentalmente en países como Estados Unidos y Canadá donde la biotecnología no tiene tan mala prensa y la tradición vitivinícola es más reciente que en el Viejo Mundo.
Hasta ahora, la diversidad genética natural de la especie parece haber ido satisfaciendo las demandas, o al menos las expectativas de la industria enológica en cuanto a la contribución de las cepas de levadura vínica a la calidad del producto final y su adaptación a las condiciones de producción. A diferencia de otros microorganismos de aplicación industrial la mejora genética de levaduras vínicas no ha sido prácticamente tomada en consideración durante decenas de años, renunciando no sólo a la ingeniería genética, sino también la mejora genética por métodos clásicos como la hibridación sexual o la mutagénesis al azar. En lo que parece un curioso movimiento de acción-reacción, parece que en los últimos años se ha despertado el interés del sector productivo por los métodos de mejora genética clásica, como alternativa a algunos éxitos de la ingeniería genética, pero tratando de evitar sus contrapartidas negativas desde el punto de vista normativo y comercial. En cualquier caso, pasado el período en que el requisito principal para una buena levadura vínica era una buena cinética de fermentación, la búsqueda de caracteres cada vez más sofisticados, relacionados con la calidad del producto, y de la combinación de varias características positivas en la misma cepa; hace que la diversidad genética natural sea cada vez más difícil de explotar con estos objetivos, y por tanto más ventajoso recurrir a estrategias de mejora genética de cepas previamente seleccionadas y con un historial de aplicación eficiente. Algunos ejemplos recientes se pueden encontrar en Ramírez et al.,18 y González et al.19 Incluso en esta recuperación de métodos genéticos tradicionales, las nuevas herramientas genómicas desarrolladas para esta especie aportan algunas ventajas, por ejemplo Marullo et al.20 han utilizado micromatrices de DNA para guiar procesos introgresión en la mejora genética por hibridación sexual de cepas de levadura vínica.
Por otra parte, la aplicación práctica de la ingeniería genética a la mejora de levaduras industriales se enfrenta a problemas técnicos objetivos, debidos en parte a la propia diversidad genética de la especie, que hace que las características de las cepas utilizadas en enología difieran en algunos aspectos relevantes de las de las cepas utilizadas en los laboratorios, por lo que muchas herramientas biotecnológicas necesitan ser adaptadas para la manipulación de estas cepas. En concreto, la transformación genética de levaduras vínicas requiere de un buen sistema para introducir el DNA recombinante en la célula receptora, sistemas para estabilizar el DNA transformante por integración en el cromosoma o por replicación autónoma, métodos para la selección de transformantes, o la identificación de los promotores más apropiados para conseguir un adecuado patrón de expresión a lo largo del proceso de fermentación. Algunos problemas como el de la introducción del DNA en las células de levadura o su estabilización se han ido resolviendo de manera más o menos fácil con ligeras modificaciones de los métodos disponibles para cepas de laboratorio. La identificación de promotores potencialmente útiles en biotecnología enológica fue abordada por primera vez por Puig et al.,21 y es un trabajo en el que las herramientas genómicas han facilitado mucho la labor, gracias a un número apreciable de trabajos publicados recientemente en los que se analiza la evolución de la expresión génica, a escala genómica de levaduras vínicas a lo largo del proceso de vinificación.
En cuanto a los métodos para la selección de transformantes, los investigadores que trabajan con cepas de laboratorio disponen de un gran número de cepas auxotrofas. En general, estos marcadores no tienen utilidad para la ingeniería genética de levaduras industriales, ya que éstas prácticamente nunca son auxotrofas, y la posibilidad de obtener un derivado auxotrofo está muy limitada por su propia estructura genómica. Si bien algunos investigadores optaron en su momento por construir un derivado auxotrofo para uridina a partir de una levadura industrial;22 el procedimiento es demasiado costoso para que se pueda aplicar de manera rutinaria a cualquier nueva cepa vínica que se pretenda transformar. La alternativa más común es utilizar marcadores dominantes de transformación. Este tipo de marcadores son normalmente genes cuya expresión permite aumentar la resistencia de la cepa a antibióticos u otros agentes antimicrobianos, sin necesidad de que la cepa que se transforma tenga previamente ningún gen defectuoso. Entre estos marcadores dominantes resulta preferible recurrir a resistencias diferentes de la resistencia a antibióticos si el objetivo es construir cepas que lleguen al mercado.23-24 Alternativamente, en ocasiones es posible eliminar de las cepas las secuencias de los marcadores, una vez se ha introducido el carácter que se deseaba.
|
Dejando aparte objetivos académicos, como el marcaje genético de las cepas con el fin de controlar la imposición de levaduras inoculadas, el aroma varietal del vino blanco fue el primer objetivo de mejora en la construcción de cepas vínicas recombinantes.25 Esto se conseguía mediante dos estrategias, la expresión en levaduras de genes que codifican enzimas que degradan polímeros de la pared celular vegetal, permitiendo una mejor extracción de los precursores; y la expresión de genes que actúan sobre los precursores glicosilados de componentes del aroma primario, favoreciendo la liberación de la aglicona.26-29 La producción de enzimas que actúan sobre componentes de la pared celular vegetal tiene el interés adicional de que puede contribuir a mejorar la extracción del mosto, y a solventar problemas en las etapas de clarificación y filtración de los vinos.30-32 Algunos autores han intentado llevar a cabo procesos de ingeniería metabólica sobre levaduras vínicas, tratando de controlar el flujo de carbono a través de diferentes rutas, para tratar de producir por ejemplo más glicerol y menos etanol,33 o aumentar la acidez, que puede ser interesante en algunas regiones.34 Los resultados de este tipo de aproximación metabólica son todavía en general poco satisfactorios. También se han diseñado levaduras para mejorar el contenido en resveratrol del vino,35 el aroma secundario,36 la tolerancia al ayuno de carbono o nitrógeno,37,38 o al estrés.39 Recientemente se han construido cepas vínicas recombinantes en las que se consigue aumentar de tres a cuatro veces la cantidad de manoproteínas del vino al final de la fermentación, entre otras potenciales ventajas, se ha demostrado que esto permite mejorar la estabilidad de esos vinos frente a quiebra proteica.40 Sin embargo, la modificación genética de levaduras vínicas que más atención atrajo desde los primeros intentos fue la construcción de levaduras capaces de llevar a cabo la fermentación maloláctica simultáneamente a la fermentación alcohólica.41-43 |
Estrategia de cotransformación
Desde hace poco tiempo existen en el mercado norteamericano (Canadá y Estados Unidos) dos levaduras vínicas recombinantes que recibieron el visto bueno de Health Canada y de la FDA, respectivamente, aunque no hay datos disponibles sobre el mercado real de las mismas. La levadura ML01 es capaz de catalizar la descarboxilación del ácido málico; de este manera se consiguen la mayor parte de los efectos positivos de la fermentación maloláctica en el vino, a la vez que se evitan muchos de los inconvenientes, empezando por la incertidumbre del momento en que ésta tendrá lugar. La levadura ECMo01 cataliza la hidrólisis de la urea,44 dado que la reacción espontánea de la urea con el etanol es cuantitativamente el mecanismo más importante de formación de carbamato de etilo en los vinos, estas levaduras permiten obtener vinos con un 90 % menos de carbamato de etilo, con el consiguiente efecto beneficioso desde el punto de vista de la protección de la salud del consumidor, así como el interés comercial dadas las restricciones a la importación de muchos países basadas en el contenido de carbamato de etilo. En ambos casos las construcciones para la superexpresión de los genes de interés están integradas en el genoma y las cepas son genéticamente estables. Pero fundamentalmente, en ninguno de los casos se conservan en el genoma de las levaduras recombinantes genes de resistencia a antibióticos, o secuencias de enterobacterias, a pesar de que se hayan utilizado en alguna fase del proceso. La eliminación de las secuencias no deseadas se ha conseguido mediante una estrategia de cotransformación.
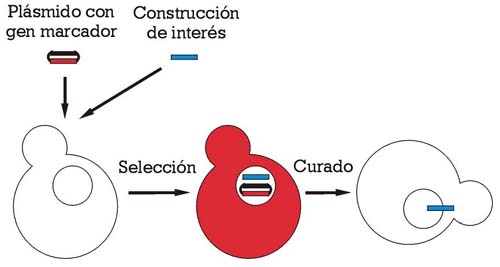
Mediante esta estrategia, ilustrada en la figura 1, se aprovecha que en los procesos de transformación de microorganismos, cuando se usa una mezcla de moléculas de DNA dos tipos diferentes, existe una probabilidad relativamente alta de que algunas de las cepas transformadas hayan incorporado moléculas de los dos tipos, de modo que la selección de transformantes con un marcador presente en una de ellas permite indirectamente seleccionar algunas que también son portadoras de la otra. En la construcción de estas levaduras el marcador de resistencia a fleomicina estaba presente en un plásmido de replicación autónoma, y la construcción de interés en un fragmento lineal que debía integrarse en el genoma. Una vez identificadas las cepas resistentes a fleomicina que eran portadoras de la construcción de interés, un simple crecimiento en ausencia de presión selectiva permitió obtener las cepas portadoras de la construcción, pero libres del plásmido.
Todos los organismos, comunidades, organizaciones, etc., están expuestos a las condiciones ambientales abióticas o bióticas. Éstas varían en intensidad, duración y complementariedad entre ellas, haciendo oscilar así los niveles de exposición. En el caso que nos ocupa, las plantas, los cultivos, pueden evitar, tolerar e incluso mejorar su adaptación a las nuevas condiciones (endurecimiento, plasticidad) (Cortina et al. 2006) con el fin de resistir esta exposición de formas muy diversas, provocando diferentes niveles de vulnerabilidad según sea su afectación (Sauri 2007). A título de ejemplo, todas las plantas están expuestas a la sequía, pero algunas mueren, otras reducen su crecimiento y otras se muestran invariables, indicando así diferentes vulnerabilidades frente a una misma exposición.
Conclusiones
Las consecuencias prácticas de la aplicación de la genómica y la ingeniería genética en enología son todavía incipientes pero prometedoras. Se puede señalar la utilización de las micromatrices de DNA para la obtención de marcadores genéticos para proceso de mejora genética de uva o levaduras vínicas. También la construcción de un número importante de levaduras vínicas transgénicas con funcionalidades de interés abre interesantes oportunidades para el futuro. En todo ello no hay que olvidar que del conocimiento surgen aplicaciones. Por ejemplo, mejorar el conocimiento de los mecanismos de adaptación de S. cerevisiae a las condiciones particulares de la vinificación, se traducirá probablemente en un futuro no muy lejano en otras aplicaciones prácticas como nuevos criterios para la selección de cepas para determinados estilos de vinificación o condiciones de fermentación, nuevos métodos de adaptación de las células de levadura, diseño de nutrientes optimizados para la fermentación, mejor control del proceso, con posibilidad de anticipar los problemas de fermentación de una manera más precisa, o control de la producción de determinados metabolitos mediante la manipulación de las condiciones de fermentación.
Bibliografía
1 Vivier, M.A., Pretorius, I.S. «Genetic improvement of grapevine: tailoring grape varieties for the Third Millenium, a review». S Afr J Enol Vitic 2000; 21: 5-26.
2 Querol, A., Fernández-Espinar; M.T., del Olmo, M., Barrio, E. «Adaptative evolution of wine yeast». Int J Food Microbiol 2003; 86: 3-10.
3 Rainieri, S., Pretorius, I.S. «Selection and improvement of wine yeasts». Ann Microbiol 2000; 50: 15-31.
4 The French-Italian Public Consortium for Grapevine Genome Characterization. Nature 2007, DOI: 10.1038/nature06148.
5 Velasco R. et al. «A high quality draft consensus sequence of the genome of a heterozygous grapevine variety». PLoS 2007 ONE 2: e1326.
6 Mezzeti, B., Pandolfini, T., Navacchi, O., Landi, L. «Genetic transformation of Vitis vinifera via organogenesis». BMC Biotechnol 2002; 2: 18-27.
7 Vidal, J.R., Kikkert, J.R., Wallace, P.G., Reisch, B.I. «High-efficiency biolistic co-transformation and regeneration of ‘Chardonnay’ (Vitis vinifera L.) containing npt-II and antimicrobial peptide genes». Plant Cell Rep 2003; 22: 252-260.
8 Vivier, M.A., Pretorius, I.S. «Genetically tailored grapevines for the wine industry». Trends in Biotechnology 2002; 20: 472-478
9 Gofieau, A., Barrell, B.G., Bussey, H., Davis, R.W., Dujon, B., Feldmann, H., Galibert, F., Hoheisel, J.D., Jacq, C., Johnston, M., Louis, E.J., Mewes, H.W., Murakami, Y., Philippsen, P., Tettelin, H., Oliver, S.G. «Life with 6000 Genes». Science 1996; 274, 546, 563-567.
10 Legras, J.L., Merdinoglu, D., Cornuet, J.M., Karst, F. «Bread, beer and wine: Saccharomyces cerevisiae diversity reflects human history». Mol Ecol 2007; 16: 2091-2102.
11 Pérez-Ortín, J.E., Querol, A., Puig, S., Barrio, E. «Molecular characterization of a chromosomal rearrangement involved in the adaptive evolution of yeast strains». Genome Res 2002; 12: 1533-1539.
12 Zuzuarregui, A., Monteoliva, L., Gil, C., del Olmo, M.L. «Transcriptomic and proteomic approach for understanding the molecular basis of adaptation of Saccharomyces cerevisiae to wine fermentation». Appl Environ Microbiol 2006; 72: 836-847.
13 Rossignol, T., Dulau, L., Julien, A., Blondin, B. «Genome-wide monitoring of wine yeast gene expression during alcoholic fermentation. Yeast 2003; 20: 1369-1385.
14 Rossignol, T., Postaire, O., Storai, J., Blondin, B. «Analysis of the genomic response of a wine yeast to rehydration and inoculation». Appl Microbiol Biotechnol 2006; 71: 699-712.
15 Beltran, G., Novo, M., Leberre, V., Sokol, S., Labourdette, D., Guillamon, J.M., Mas, A., Francois, J., Rozes, N. «Integration of transcriptomic and metabolic analyses for understanding the global responses of low-temperature winemaking fermentations». FEMS Yeast Res 2006; 6: 1167-1183.
16 Marks, V.D., Sui, SJH., Erasmus, D., van der Merwe, G.K., Brumm., Wasserman, W.W., Bryan, J., van Vuuren, H.J.J. «Dynamics of the yeast transcriptome during wine fermentation reveals a novel fermentation stress response». FEMS Yeast Res 2008; 8: 35-52.
17 Varela, C., Cardenas, J., Melo, F., Agosin, E. «Quantitative analysis of wine yeast gene expression profiles under winemaking conditions». Yeast 2005; 22: 369-383.
18 Ramírez, M., Regodón, J.A, Pérez, F. Rebollo, J.E. «Wine yeast fermentation vigor may be improved by elimination of recessive growth-retarding alleles».Biotechnol Bioen 1999; 65: 212-218.
19 González, R., Martínez-Rodríguez, A., Carrascosa, A. «Yeast autolytic mutants potentially useful for sparkling wine production». Int J Food Microbiol 2003; 84: 21-26.
20 Marullo, P., Yvert, G., Bely, M., Aigle, M., Dubourdieu D. «Efficient use of DNA molecular markers to construct industrial yeast strains». FEMS Yeast Res 2007; 7: 1295-1306.
21 Puig, S., Querol, A., Ramón, D., Pérez-Ortín, J.E. «Evaluation of the use of phase-specific gene promoters for the expression of enological enzymes in an industrial wine yeast strain». Biotechnol Letters 1996; 18: 887-892.
22 Puig, S., Ramón, D., Pérez-Ortín, J.E. «Optimized method to obtain stable food-safe recombinant wine yeast strains». J Agric Food Chem 1998; 46: 1689-1693.
23 Xie, Q., Jiménez, A. «Molecular cloning of a novel allele of SMR1 which determines sulfometuron methyl resistance in Saccharomyces cerevisiae». FEMS Microbiol Letters 1996; 137:165-168.
24 Cebollero, E., González, R. «Comparison of two alternative dominant selectable marker for wine yeast transformation». Appl Environ Microbiol 2004; 70: 7018-7023.
25 Pérez-González, J.A, González, R., Querol, A., Sendra, J., Ramón, D. «Construction of a recombinant wine yeast strain expressing a β-(1,4)-endoglucanase activity and its use in microvinification processes». Appl Environ Microbiol 1993; 59: 2801-2806.
26 Ganga, M.A., Piñaga, F., Vallés, S., Ramón, D., Querol, A. «Aroma improving in microvinification processes by the use of a recombinant wine yeast strain expressing the Aspergillus nidulans xlnA gene». Int J Food Microbiol 1999; 47: 171-178.
27 Manzanares, P., Orejas, M., Gil, J.V, de Graaf, L., Visse, J., Ramón, D. «Construction of a genetically modified wine yeast strain expressing the Aspergillus aculeatus rhaA gene, encoding an α-L-rhamnosidase of enological interest». Appl Environ Microbiol 2003; 69: 7558-7562.
28 Sánchez-Torres, P., González-Candelas, L., Ramón, D. «Expression on a wine yeast strain of the Aspergillus niger abfB gene». FEMS Microbiol Letters 1996; 145: 189-194.
29 Sánchez-Torres, P., González-Candelas, L., Ramón, D. «Heterologous expression of a Candida molischiana anthocyanin-β-glucosidase in a wine yeast strain». J Agric Food Chem 1998; 46: 354-360.
30 González-Candelas, L., Cortell, A., Ramón, D. «Construction of a recombinant wine yeast strain expressing a fungal pectate lyase gene». FEMS Microbiol Letters 1995; 126: 263-269.
31 Vilanova, M., Blanco, P., Cortés, S. Cortés, S., Castro, M., Villa, T.G. «Use of a PGU1 recombinant Saccharomyces cerevisiae strain in oenological fermentations».J Appl Microbiol 2000; 89: 876-883.
32 Fernández-González, M., Úbeda, J.F., Cordero-Otero, R.R. Thanvanthri Gururajan, V., Briones, A.I. Engineering of an oenological Saccharomyces cerevisiae strain with pectinolytic activity and its effect on wine». Int J Food Microbiol 2005; 102: 173-183.
33 Michnick, S., Roustanm, J.L., Remize, F., Barre, P., Dequin, S. «Modulation of glycerol and ethanol yields during alcoholic fermentation in Saccharomyces cerevisiae strains overexpressed or disrupted for GPDI encoding glycerol 3-phosphate dehydrogenase». Yeast1997; 13: 783-793.
34 Dequin, S., Barre, P. «Mixed lactic acid-alcoholic fermentation by Saccharomyces cerevisiae expressing the Lactobacillus casei L(+)-LDH». Biotechnology (NY) 1994; 12: 173-177.
35 González-Candelas, L., Gil, J.V, Lamuela-Raventós, R.M., Ramón, D. «The use of transgenic yeasts expressing a gene encoding a glycosyl-hydrolase as a tool to increase resveratrol content in wine». Int J Food Microbiol 2000; 59: 179-183.
36 Lilly, M., Lambrechts, M.G,, Pretorius. I.S. «Effect of increased yeast alcohol acetyltransferase activity on flavor profiles of wine and distillates». Appl Environ Microbiol 2000; 66: 744-753.
37 Pérez-Torrado, R., Gimeno-Alcañiz, J.V, Matallana, E. «Wine yeast strains engineered for glycogen overproduction display enhanced viability under glucose deprivation conditions». Appl Environ Microbiol 2002; 68: 3339-3344.
38 Salmon, J.M., Barre, P. «Improvement of nitrogen assimilation and fermentation kinetics under enological conditions by derepression of alternative nitrogen-assimilatory pathways in an industrial Saccharomyces cerevisiae strain». Appl Environ Microbiol 1998; 64: 3831-3837.
39 Cardona, F., Carrasco, P., Pérez-Ortín, J.E., del Olmo, M.L., Aranda, A. «A novel approach for the improvement of stress resistance in wine yeasts». Int J Food Microbiol 2007; 114: 83-91.
40 González-Ramos, D., Cebollero, E., González, R. «A recombinant wine yeast strain overproducing mannoproteins stabilizes wine against protein haze». Appl Environ Microbiol 2008 (en prensa).
41 Volschenk, H.., Viljoen, M., Grobler, J., Bauer, F., Lonvaud-Funel, A., Denayrolles, M., Subden, R.E., van Vuuren, H.J.J. «Malolactic fermentation in grape musts by a genetically engineered strain of Saccharomyces cerevisiae». Am J Enol Vitic 1997a; 48: 193-197.
42 Volschenk, H., Viljoen, M., Grobler, J., Petzold, B., Bauer, F., Subden, R.E., Young, R.A., Lonvaud, A., Denayrolles, M., van Vuuren, HJJ. «Engineering pathways for malate degradation in Saccharomyces cerevisiae». Nature Biotechnol 1997b; 15: 253-257.
43 Husnick, J.I, Volschenk, H., Bauer, J. Covalizza, D., Luo, Z., van Vuuren, H.J.J. «Metabolic engineering of malolactic wine». Yeast Metab Eng 2006; 8: 315-323.
44 Coulon, J., Husnik, JI., Inglis, D.L., van der Merwe, G.K., Lonvaud, A., Erasmus, D.J., van Vuuren, H.J.J. «Metabolic engineering of Saccharomyces cerevisiae to minimize the production of ethyl carbamate in wine». Am J Enol Vitic 2006; 57: 113-124.