Resumen
Los mercaptanos polifuncionales, como los tioles varietales, son compuestos clave en el perfil aromático de vinos blancos. Estos compuestos, asociados a aromas tropicales y herbáceos, se encuentran en las uvas en forma de precursores no volátiles que requieren la acción enzimática de levaduras durante la fermentación para liberar la fracción volátil y, en consecuencia, los aromas asociados. Factores como la variedad de uva, las prácticas vitícolas y las condiciones ambientales influyen directamente en la cantidad de precursores disponibles y en su transformación en aromas volátiles durante la vinificación.
El metabolismo de las levaduras, en particular su capacidad para internalizar y procesar estos precursores es fundamental para determinar el contenido final de tioles en el vino. Variaciones genéticas entre levaduras y diferencias en las prácticas enológicas pueden potenciar o limitar la liberación de estos compuestos aromáticos. Estrategias biotecnológicas, como la selección de cepas con rasgos genéticos determinados o su modificación mediante técnicas de mejora genética, han demostrado su eficacia para incrementar la concentración de tioles en los vinos. Además, el uso de consorcios microbianos y nuevas tecnologías como la biología sintética ofrecen perspectivas prometedoras para mejorar la producción de tioles, manteniendo un balance óptimo entre capacidad fermentativa y generación de aromas. A medida que la industria enfrenta desafíos como el cambio climático y los cambios en las preferencias del consumidor, el desarrollo de estas soluciones innovadoras promete una mejora significativa en la calidad sensorial de los vinos.
Introducción
Los mercaptanos polifuncionales son compuestos aromáticos sulfurados que juegan un papel clave en la diferenciación sensorial de los vinos, principalmente en blancos y especialmente aquellos elaborados a partir de variedades como sauvignon blanc, semillón o verdejo. Entre ellos, los tioles varietales contribuyen significativamente a la calidad aromática de los vinos, aportando aromas característicos a frutas tropicales y aromas herbáceos, lo que los convierte en uno de los aspectos más estudiados en la química del vino en las últimas décadas (Ruiz et al., 2019). A pesar de los avances en la comprensión de la base genética y metabólica de la síntesis de los precursores de tioles varietales en la vid, y de su revelación por levaduras, todavía quedan muchas incógnitas por resolver. La identificación de los genes responsables de la producción de precursores de tioles en la vid y cómo se regula su expresión en respuesta a diferentes estímulos ambientales, son áreas que requieren más investigación. Asimismo, la elucidación de las variantes genéticas entre las distintas variedades de uva podría ofrecer nuevas herramientas para la selección de cultivares con un mayor potencial para la producción de vinos aromáticos ricos en tioles.
Los principales compuestos que conforman la fracción varietal tiólica de los vinos son el 4MSP (4-metil-4-sulfanilpentan-2-ona), el 3MH (3-mercaptohexan-1-ol) y su derivado acetilado, el 3MHA (acetato de 3-mercaptohexilo) que, por su bajo umbral de percepción, tienen un gran impacto sensorial en el perfil aromático del vino. Estos tioles no se encuentran en su forma volátil en las uvas, sino que están presentes como precursores conjugados con aminoácidos u oligopéptidos (principalmente cisteína y glutatión) que requieren la acción de enzimas de las levaduras durante la fermentación para liberar los compuestos volátiles responsables del aroma. Sin embargo, el proceso de liberación de tioles no es solo una cuestión de la capacidad enzimática de las levaduras, sino que también está influido por la cantidad y la disponibilidad de precursores en el mosto de uva. La disponibilidad de estos precursores en el mosto depende, además de la variedad de uva, de las prácticas vitícolas y las condiciones ambientales (Kobayashi et al., 2011), así como de factores bióticos como la infección por Botrytis cinerea (Thibon et al., 2011). Estos estímulos pueden inducir la expresión de ciertos genes, aumentando la acumulación de precursores de tioles en las bayas, lo que afecta directamente a la capacidad de las levaduras para liberar los tioles durante la fermentación.
El proceso de liberación de tioles durante la fermentación depende de las levaduras, capaces de internalizar sus precursores y expresar enzimas con actividad β-liasa capaces de romper los conjugados de cisteína y glutatión liberando los tioles volátiles responsables del aroma del vino (Roncoroni et al., 2011). Además de S. cerevisiae, otras especies de levadura presentan capacidad para liberar tioles, y han demostrado ser herramientas útiles para incrementar la concentración de estos compuestos en el vino, bien por disponer de variantes genéticas sobre los genes implicados (Belda et al., 2017a), bien por contar con una regulación transcripcional distinta a la de S. cerevisiae (Ruiz et al., 2020) o bien por desencadenar procesos de interacción interespecífica que den lugar a efectos sinérgicos entre las levaduras activas durante la fermentación (Renault et al., 2016). Sin embargo, se ha observado que, aunque las levaduras tienen la capacidad de liberar tioles, solo una pequeña fracción de los precursores presentes en el mosto es efectivamente transformada en compuestos volátiles. Esto se debe en parte a la compleja regulación de la expresión de los genes involucrados en la internalización y posterior liberación de tioles en levaduras, y pone de manifiesto la necesidad de seguir comprendiendo las bases moleculares de la liberación de tioles en vinificación, para desarrollar herramientas microbiológicas o itinerarios enológicos que permitan maximizar su liberación.
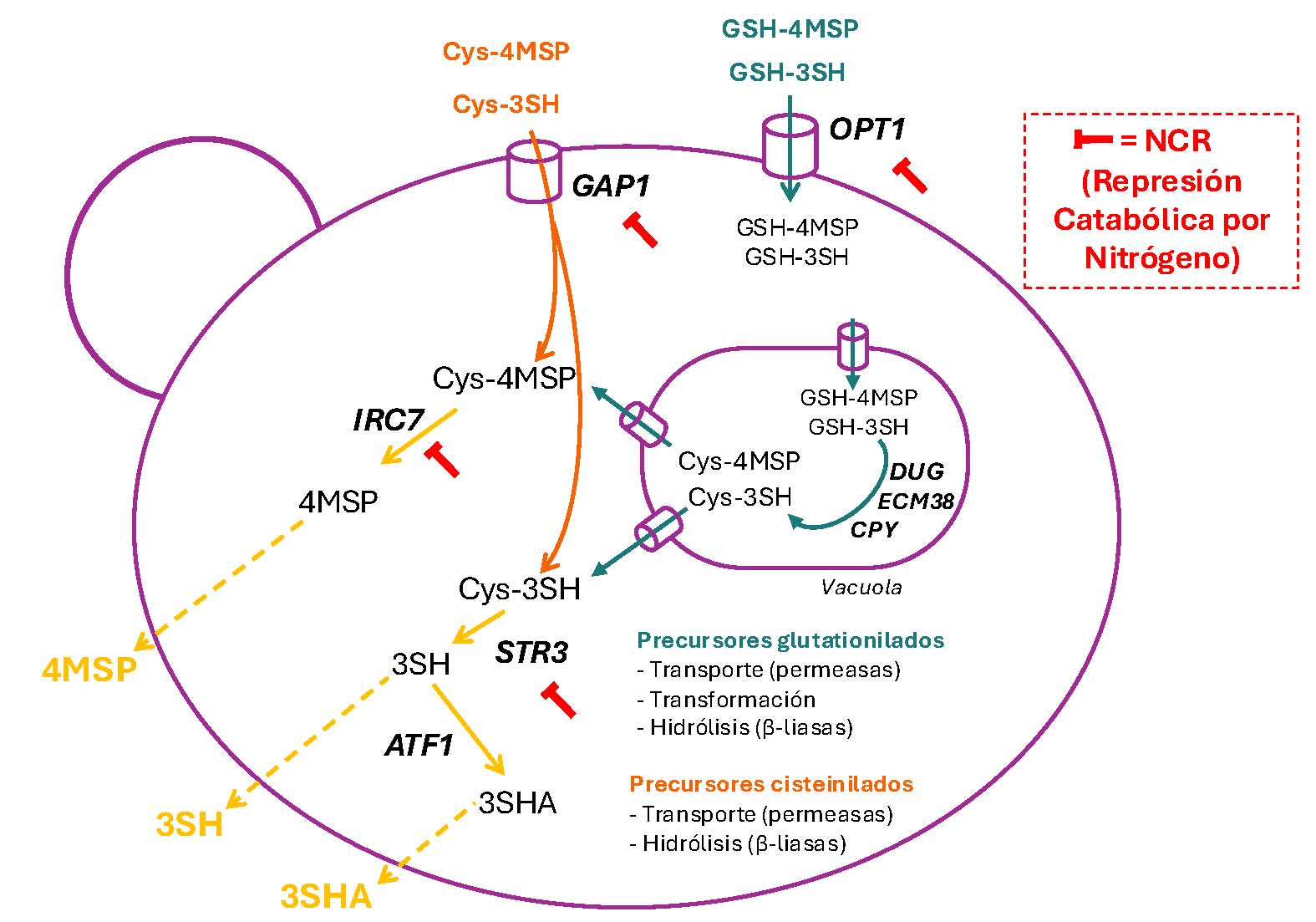
Bases genéticas de la liberación de tioles en levaduras
El contenido final de tioles en el vino depende, en primera instancia, de la presencia y concentración de sus precursores en el mosto, pero el metabolismo de las levaduras durante la fermentación es también un aspecto clave en la liberación de aromas tiólicos. Las levaduras, tanto S. cerevisiae como otras especies no-Saccharomyces, internalizan los precursores tiólicos y los hidrolizan enzimáticamente, utilizando el amonio y el piruvato derivados de esta hidrólisis para su metabolismo, y liberando la fracción volátil del precursor en forma de tioles libres (Howell et al., 2005). En S. cerevisiae, se conocen los genes implicados en este proceso (Santiago & Gardner, 2015) (Figura 1). Los precursores tiólicos (principalmente: cys-3SH, GSH-3SH y cys-4MSP) son internalizados mediante transportadores generales de aminoácidos y oligopéptidos. OPT1 se ha descrito como el principal transportador de los precursores glutationilados, mientras que el transportador general de aminoácidos GAP1 se ha descrito como responsable de la mayor parte de la internalización de precursores cisteinilados (Subileau et al. 2008). Una vez en el citoplasma, enzimas con actividad β-liasa rompen el enlace C-S de los precursores cisteinilados. Los genes BNA3, CYS3, GLO1 y, principalmente, IRC7 codifican las enzimas responsables de la liberación de 4MSP a partir de Cys-4MSP (Howell et al., 2005; Roncoroni et al., 2011). Por otro lado, el gen STR3 ha sido identificado como responsable de la liberación de 3SH a partir de Cys-3SH, aunque cabe destacar su baja especificidad por este sustrato (Holt et al., 2012). Respecto a los precursores glutationilados, una vez internalizados, se transforman en precursores cisteinilados a través de una compleja ruta en la vacuola que involucra transportadores vacuolares (gen YBT1) y enzimas codificadas por los genes DUG1, DUG2, DUG3, ECM38, entre otros, y posteriormente siguen las vías de hidrólisis catalizadas por enzimas con actividad β-liasa (Belda et al., 2017b) (Figura 1). En cuanto al tiol 3SHA, el derivado acetilado de 3SH, su formación a partir de 3SH está catalizada por la enzima codificada por el gen ATF1 (Swiegers et al., 2006).
El conocimiento sobre los determinantes genéticos de la producción de tioles en levaduras ha impulsado la exploración de variantes genéticas en los genes involucrados en este metabolismo, especialmente en IRC7. Roncoroni et al. (2011) describieron la existencia de dos variantes alélicas de este gen en S. cerevisiae: una variante completa (IRC7F) y otra portadora de una deleción de 38 pb (IRC7S). Esta deleción altera el marco abierto de lectura, generando un codón stop temprano que produce una enzima más corta (340 aa en lugar de 400 aa) con menor actividad catalítica. Estudios posteriores realizados por nuestro grupo confirmaron que las cepas de S. cerevisiae homocigotas para el alelo IRC7S presentan actividad β-liasa reducida, lo que resulta en una producción baja o nula del tiol 4MSP (Belda et al., 2016). Además, ambos estudios mostraron que la mayoría de las cepas vínicas de S. cerevisiae (cerca de un 90 %) son homocigotas para el alelo delecionado IRC7S. Esta situación algo contraintuitiva, en la que la variante alélica menos funcional de IRC7 se encuentra ampliamente distribuida en las cepas vínicas, se ha explicado mediante la asociación del alelo IRC7S con particularidades fenotípicas y genómicas que favorecen su prevalencia en hábitats domesticados, especialmente en el vino (Ruiz et al., 2021). Cordente et al. (2019) demostraron además que la deleción previamente descrita del gen IRC7 no explica completamente las diferencias en la actividad de la enzima Irc7p. Identificaron varios polimorfismos de un solo nucleótido en la secuencia de IRC7 que, junto con la longitud del alelo, explican de forma más precisa las diferencias en la actividad de esta enzima y, por tanto, en la capacidad de liberar tioles entre cepas de S. cerevisiae.
Aunque el impacto de las variantes genéticas en los genes de transportadores de precursores tiólicos, como OPT1 o GAP1, en la producción de tioles no ha sido explorado en profundidad, se han identificado diversas familias de transportadores de oligopéptidos en diferentes cepas de S. cerevisiae. Estas familias muestran preferencias específicas por péptidos de distintos tamaños (como los precursores tiólicos) y podrían representar una ventaja adaptativa en ambientes con baja disponibilidad de nitrógeno, como el mosto de uva (Becerra-Rodriguez et al., 2020). Asimismo, existen diferencias en la preferencia de distintas especies por distintos tipos de precursores tiólicos presentes en el mosto de uva, lo que determina su contribución directa al perfil de aromas tiólicos en los vinos (Vicente & Kiene et al., 2024). Futuros estudios deberán determinar los determinantes genéticos (variantes alélicas, variación en el número de copias de genes, etc.) que justifican la capacidad de distintas especies para transportar precursores tiólicos, así como su respuesta a mecanismos de regulación transcripcional, claves en este proceso, como la Represión Catabólica por Nitrógeno (NCR) (Berg et al., 2023).
Regulación de la expresión de los genes involucrados en la liberación de tioles
Como ya se ha comentado, ciertos transportadores localizados en la membrana plasmática juegan un papel esencial en la captación de precursores tiólicos desde el medio extracelular (mosto de uva) hacia el interior de las células de levadura. La expresión de los principales genes involucrados (OPT1 -en menor medida, OPT2– y GAP1) está regulada por la disponibilidad de nutrientes nitrogenados en el medio, a través de mecanismos de NCR. Este mecanismo de regulación depende de la actuación de factores de transcripción como Gat1p y Gln3p, que inducen la expresión de genes relacionados con el metabolismo de fuentes no preferenciales de nitrógeno. Estos factores de transcripción están a su vez regulados por la proteína Ure2p, activa en condiciones de disponibilidad de fuentes preferenciales de nitrógeno (i.e. amonio y algunos aminoácidos como la glutamina) y cuya presencia bloquea la acción de Gat1p y Gln3p, inhibiendo la transcripción de genes que codifican transportadores y enzimas involucradas en el catabolismo de fuentes menos preferenciales de nitrógeno, como son los aminoácidos y oligopéptidos que se encuentran conjugados con los aromas tiólicos (Thibon et al., 2008). Cuando la disponibilidad de fuentes preferenciales de nitrógeno disminuye, Gln3p y Gat1p son liberados y se translocan al núcleo, activando la expresión de genes para el metabolismo de fuentes alternativas de nitrógeno. Asimismo, IRC7, principal gen responsable de la producción de enzimas con actividad β-liasa sobre los precursores del 4-MSP, está regulado por NCR, y por tanto inhibida su expresión en condiciones de alta disponibilidad de fuentes preferenciales de nitrógeno en el mosto (Thibon et al., 2008). Además, en este caso, la disponibilidad de azufre y de cobre en el medio juegan también un papel crucial en regular la expresión de las enzimas con actividad β-liasa, como CYS3, cuya expresión se dispara en condiciones de déficit de azufre en el medio (Boer et al., 2003), o IRC7, cuya expresión disminuye ante concentraciones elevadas de cobre (Gross et al., 2000).
Cabe destacar que los niveles de expresión génica, además de responder a factores ambientales, varían entre cepas de levadura, lo que, junto con la existencia de variantes alélicas puede explicar la distinta capacidad de distintas cepas o especies para liberar tioles en fermentación (Tofalo et al., 2020). Asimismo, distintas cepas de levadura presentan distintos grados de sensibilidad a los mecanismos de NCR, aspecto que también se ha demostrado clave en la explicación de los distintos niveles de liberación de tioles volátiles entre cepas (Deed et al. 2011; Dufour et al., 2013). De esta forma, en las últimas dos décadas se han descrito los mecanismos moleculares que determinan la liberación de compuestos tiólicos en fermentaciones vínicas y se han definido los aspectos clave a tener en cuenta en procesos de bioprospección de cepas de S. cerevisiae con características enológicas óptimas, explotando la diversidad natural de la especie.
Aproximaciones biotecnológicas para la mejora de la liberación de tioles
Gracias a todo el conocimiento expuesto anteriormente, en la actualidad se dispone de cepas de levaduras con una capacidad elevada para la liberación de tioles varietales en vinos, y se conocen ciertas estrategias enológicas necesarias para maximizarla (i.e. optimización de la nutrición nitrogenada). A pesar de ello, la biotecnología microbiana ha puesto sobre la mesa herramientas alternativas que pueden contribuir a la mejora de la eficiencia de este proceso. Estas incluyen el empleo de ingeniería genética, el empleo de consorcios microbianos o la biología sintética como herramientas clave para potenciar la producción de compuestos aromáticos tiólicos.
El empleo de técnicas de ingeniería genética está regulado por normativas específicas que limitan su aplicación industrial, aunque, a nivel experimental, se han desarrollado decenas de mutantes para la mejor comprensión de los determinantes genéticos de la liberación de tioles. El primer trabajo que empleó ingeniería genética para incrementar la liberación de tioles en vinos introdujo el gen tnaA de la bacteria Escherichia coli, que codifica una triptofanasa con actividad cistationina β-liasa, en S. cerevisiae de dos formas distintas: integrándolo en el genoma de la levadura, y en forma un plásmido multicopia (extracromosómico) (Swiegers et al., 2007). Los resultados mostraron que la integración del gen en el genoma incrementaba la producción de los tioles volátiles 4MSP y 3SH en concentraciones hasta 25 veces superiores respecto a las cepas no modificadas, y mayores que las obtenidas con plásmidos multicopia, destacando de esta forma la importancia de la estabilidad del gen y su expresión en la actividad enzimática para la liberación de tioles. En trabajos posteriores, Graf y Buchhaupt (2023) emplearon un enfoque similar para insertar tnaA, pero esta vez acompañado de genes propios de S. cerevisiae implicados en el metabolismo del glutatión, como GSH1 (glutatión sintasa) y GTT1 (glutatión S-transferasa) para incrementar el metabolismo del glutatión por parte de la levadura. Así, la combinación de ambos genes junto con tnaA multiplicó por 10 la producción de 3SH comparado con la cepa portadora únicamente del gen tnaA. Además, durante el siglo XXI se han desarrollado técnicas de clonación extremadamente versátiles y eficaces. Herramientas de edición génica como CRISPR-Cas9 (y similares), se han empleado también para la edición genética de S. cerevisiae. Por ejemplo, se han generado mutantes para incrementar la producción de 3SH gracias a la introducción del gen tnaA. En este caso, la introducción del gen tnaA lleva asociada el incremento de forma natural en la levadura de la actividad de genes nativos codificantes para las acetil transferasas ATF1 y ATF2, enzimas que transforman el 3SH en su derivado acetilado, 3-SHA. Estas cepas alcanzaron concentraciones finales de hasta 7.000 ng/L de 3-SHA en sauvignon blanc (100 veces más que en la fermentación control), intensificando el perfil aromático del vino (Kiene et al., 2021).
No obstante, existen otras técnicas, como la mutagénesis al azar combinada con enfoques de genética clásica, que han permitido desarrollar cepas con características optimizadas compatibles con sus aplicaciones industriales y que cumplen con la normativa vigente y los requisitos de la industria. En relación con estos enfoques, se han realizado estudios que, empleando molecular breeding, un método que combina la reproducción y recombinación sexual entre cepas comerciales de S. cerevisiae y cepas portadoras de mutaciones específicas (como en el gen URE2), han obtenido resultados prometedores en cuanto al incremento de la producción de tioles. De este modo, mediante el cruce de cepas con mutaciones beneficiosas en URE2 pero con baja aptitud enológica con cepas comerciales, seguido del retrocruzamiento de sus descendientes con el parental comercial durante cuatro generaciones, se generaron cepas de S. cerevisiae que mantenían las características tecnológicas óptimas de la cepa comercial y, al mismo tiempo, portaban la mutación en el gen URE2 procedente de la cepa no comercial. Esto permitió incrementar la concentración de tioles volátiles entre 2 y 7 veces respecto a la fermentación control (Dufour et al., 2013).
Por otro lado, el empleo de cepas seleccionadas de distintas especies no-Saccharomyces también ha mostrado resultados relevantes en la producción de tioles en fermentaciones vínicas. Especies como Metschnikowia pulcherrima y Torulaspora delbrueckii destacan por su capacidad para liberar 3SH y su derivado acetilado, dependiendo de la cepa y las condiciones de fermentación. Distintos trabajos han demostrado que su uso en fermentaciones secuenciales junto con S. cerevisiae incrementan la liberación de tioles (Zott et al., 2011; Belda et al., 2017a; Ruiz et al., 2018). También se ha descrito recientemente el papel positivo de Lachancea thermotolerans en la liberación de tioles, como consecuencia de una mayor capacidad de consumo y transformación de los precursores tiólicos, especialmente 3-SH a partir de su precursor glutationilado (Vicente & Kiene et al., 2024). En esta línea, cabe destacar el diseño de consorcios microbianos complejos (con múltiples especies) que explotan distintos rasgos de S. cerevisiae y especies no-Saccharomyces para optimizar la liberación de compuestos aromáticos manteniendo una cinética fermentativa adecuada. Albertin et al. (2017) combinaron cinco especies distintas: M. pulcherrima y T. delbrueckii para incrementar la liberación de tioles, Hanseniaspora uvarum y Candida zemplinina para retrasar el crecimiento de S. cerevisiae y así favorecer la acción de las dos primeras, y S. cerevisiae, para finalizar de manera adecuada el proceso fermentativo. Este tipo de aproximaciones no están exentas de complicaciones en su práctica industrial, tanto por la complejidad de producir inóculos industriales de múltiples especies, como por el posterior control del comportamiento de inóculos complejos. En ese sentido, hemos podido demostrar que el comportamiento de consorcios complejos (conteniendo de 2 a 6 especies), además de presentar propiedades emergentes que pueden dar lugar a nuevas funciones metabólicas de interés industrial, es predecible a partir del conocimiento de la ecología de las distintas especies que forman parte de dicho consorcio (Ruiz et al., 2023), por lo que el uso de consorcios microbianos en enología es prometedor.
Finalmente, a futuro, la biología sintética ofrece un enfoque prometedor y podemos anticipar que esta disciplina permitirá, en el marco de la industria enológica, rediseñar las cepas de levadura actuales o crear sistemas biológicos completamente nuevos con el objetivo de optimizar la calidad sensorial de los vinos. Estos sistemas, deberán combinar las características propias de S. cerevisiae, como su elevada capacidad fermentativa, con las de otras especies no-Saccharomyces, que gracias a su metabolismo y su gran batería enzimática incrementen la complejidad sensorial de los vinos (Belda et al., 2021; Walker & Pretorius, 2022). De esta forma, se generarán cepas quiméricas, más versátiles en cuanto a producción aromática, pero que mantengan un perfil fermentativo robusto frente a los desafíos actuales a los que se enfrenta la industria; desafíos derivados principalmente del cambio en las condiciones climáticas (vinos con grados alcohólicos probables elevados, deficitarios en nutrientes y con desbalances en cuanto a su acidez) y las demandas del consumidor.
Bibliografía
Albertin, W., Zimmer, A., Miot-Sertier, C., Bernard, M., Coulon, J., Moine, V., Colonna-Ceccaldi, B., Beli, M., Marullo, P., y Masneuf-Pomarede, I.: “Combined effect of the Saccharomyces cerevisiae lag phase and the non-Saccharomyces consortium to enhance wine fruitiness and complexity”. Applied microbiology and biotechnology, 2017, 101, 7603-7620.
Becerra-Rodríguez, C., Marsit, S., y Galeote, V.: “ Diversity of oligopeptide transport in yeast and its impact on adaptation to winemaking conditions”. Frontiers in Genetics, 2020, 11, 602.
Belda, I., Ruiz, J., Navascués, E., Marquina, D., y Santos, A.: “ Improvement of aromatic thiol release through the selection of yeasts with increased β-lyase activity”. International Journal of Food Microbiology, 2016, 225, 1-8.
Belda, I., Ruiz, J., Beisert, B., Navascués, E., Marquina, D., Calderón, F., Rauhut, D., Benito, S. y Santos, A.: “Influence of Torulaspora delbrueckii in varietal thiol (3-SH and 4-MSP) release in wine sequential fermentations”. International Journal of Food Microbiology, 2017a, 257, 183-191.
Belda, I., Ruiz, J., Esteban-Fernández, A., Navascués, E., Marquina, D., Santos, A., y Moreno-Arribas, M. V.: “Microbial contribution to wine aroma and its intended use for wine quality improvement”. Molecules, 2017b, 22(2), 189.
Belda, I., Williams, T. C., de Celis, M., Paulsen, I. T., y Pretorius, I. S.: “Seeding the idea of encapsulating a representative synthetic metagenome in a single yeast cell”. Nature Communications, 2021, 12(1), 1599.
Berg, H. Y., Arju, G., Becerra-Rodríguez, C., Galeote, V., & Nisamedtinov, I.: “Unlocking the secrets of peptide transport in wine yeast: insights into oligopeptide transporter functions and nitrogen source preferences”. Applied and Environmental Microbiology, 2023, 89(11), e01141-23.
Boer, V.M., de Winde, J.H., Pronk, J.T., y Piper, M.D.: “The genome-wide transcriptional responses of Saccharomyces cerevisiae grown on glucose in aerobic chemostat cultures limited for carbon, nitrogen, phosphorus, or sulfur”. Journal of Biological Chemistry, 2003, 278(5):3265-3274.
Cordente, A. G., Borneman, A. R., Bartel, C., Capone, D., Solomon, M., Roach, M., y Curtin, C. D.: “Inactivating mutations in Irc7p are common in wine yeasts, attenuating carbon-sulfur β-lyase activity and volatile sulfur compound production”. Applied and Environmental Microbiology, 2019, 85(6), e02684-18.
Deed, N.K., van Vuuren, H.J., y Gardner, R.C.: “Effects of nitrogen catabolite repression and di-ammonium phosphate addition during wine fermentation by a commercial strain of S. cerevisiae”. Applied Microbiology and Biotechnology, 2011, 89(5):1537–1549.
Dufour, M., Zimmer, A., Thibon, C., y Marullo, P.: “Enhancement of volatile thiol release of Saccharomyces cerevisiae strains using molecular breeding”. Applied Microbiology and Biotechnology, 2013, 97, 5893-5905.
Graf, F.M.R., y Buchhaupt, M.: “Engineering volatile thiol formation in yeast”. Journal of Applied Microbiology, 2023, 134(2) lxac078.
Gross, C., Kelleher, M., Iyer, V. R., Brown, P. O., y Winge, D. R.: “Identification of the copper regulon in Saccharomyces cerevisiae by DNA microarrays”. Journal of Biological Chemistry, 2000, 275, 32310–32316.
Holt, S., Cordente, A. G., y Curtin, C.: “Saccharomyces cerevisiae STR3 and yeast cystathionine β-lyase enzymes: The potential for engineering increased flavor release”. Bioengineered Bugs, 2012, 3(3), 180-182.
Howell, K. S., Klein, M., Swiegers, J. H., Hayasaka, Y., Elsey, G. M., Fleet, G. H., Høj, P. B., Pretorius, I.S., y de Barros Lopes, M. A.: “Genetic determinants of volatile-thiol release by Saccharomyces cerevisiae during wine fermentation.” Applied and Environmental Microbiology, 2005, 71(9), 5420-5426.
Kiene, F., Pretorius, I. S., Rauhut, D., von Wallbrunn, C., y van Wyk, N.: “Construction and analysis of a yeast for the simultaneous release and esterification of the varietal thiol 3-sulfanylhexan-1-ol”. Journal of Agricultural and Food Chemistry, 2021, 69(40), 11919-11925.
Kobayashi, H. Takase, H., Suzuki, Y., Tanzawa, F., Takata, R., Fujita, K., Kohno, M., Mochizuki, M., Suzuki, S., y Konno, T.: “ Environmental stress enhances biosynthesis of flavor precursors, S-3-(hexan-1-ol)-glutathione and S-3-(hexan-1-ol)-L-cysteine, in grapevine through glutathione S-transferase activation.”.Journal of Experimental Botany, 2011, 62, 1325–1336.
Renault, P., Coulon, J., Moine, V., Thibon, C., y Bely, M.: “Enhanced 3-Sulfanylhexan-1-ol Production in Sequential Mixed Fermentation with Torulaspora delbrueckii/Saccharomyces cerevisiae Reveals a Situation of Synergistic Interaction between Two Industrial Strains”. Frontiers in Microbiology, 2016, 7:293.
Roncoroni, M., Santiago, M., Hooks, D. O., Moroney, S., Harsch, M. J., Lee, S. A., Richards, K.D., Nicolau, L., y Gardner, R. C.: “The yeast IRC7 gene encodes a β-lyase responsible for production of the varietal thiol 4-mercapto-4-methylpentan-2-one in wine”. Food Microbiology, 2011, 28(5), 926-935.
Ruiz, J., Belda, I., Beisert, B., Navascués, E., Marquina, D., Calderón, F., Rauhut, D., Santos, A., y Benito, S.: “Analytical impact of Metschnikowia pulcherrima in the volatile profile of Verdejo white wines”. Applied microbiology and biotechnology, 2018, 102, 8501-8509.
Ruiz, J., Kiene, F., Belda, I., Fracassetti, D., Marquina, D., Navascués, E., Calderón, F., Benito, A., Rauhut, D., Santos, A., & Benito, S.: “Effects on varietal aromas during wine making: a review of the impact of varietal aromas on the flavor of wine”. Applied Microbiology and Biotechnology 2019, 103, 7425–7450.
Ruiz, J., de Celis, M., de Toro, M., Mendes-Ferreira, A., Rauhut, D., Santos, A., y Belda, I.: “Phenotypic and transcriptional analysis of Saccharomyces cerevisiae during wine fermentation in response to nitrogen nutrition and co-inoculation with Torulaspora delbrueckii.” Food Research International, 2020, 137, 109663.
Ruiz, J., de Celis, M., Martín‐Santamaría, M., Benito‐Vázquez, I., Pontes, A., Lanza, V. F., Sampaio, J.P., Santos, A., y Belda, I.: “Global distribution of IRC7 alleles in Saccharomyces cerevisiae populations: a genomic and phenotypic survey within the wine clade”. Environmental Microbiology, 2021, 23(6), 3182-3195.
Ruiz, J., de Celis, M., Vila J., Diaz-Colunga, J., Benitez-Dominguez, B., Vicente, J., Santos, A., Sanchez, A., y Belda I.: “Predictability of the community-function landscape in wine yeast ecosystems”. Molecular Systems Biology, 2023, 19: e11613.
Santiago, M., y Gardner, R. C.: “Yeast genes required for conversion of grape precursors to varietal thiols in wine”. FEMS Yeast Research, 2015, 15(5), fov034.
Subileau, M., Schneider, R., Salmon, J-M., y Degryse, E.: “Nitrogen catabolite repression modulates the production of aromatic thiols characteristic of Sauvignon Blanc at the level of precursor transport”. FEMS Yeast Research, 2008, 8:771–780.
Swiegers, J. H., Willmott, R., Hill-Ling, A., Capone, D. L., Pardon, K. H., Elsey, G. M., Howell, K.S., de Barros Lopes, M.A., Sefton, M.A., Lilly, M., y Pretorius, I. S.: “Modulation of volatile thiol and ester aromas by modified wine yeast”. Developments in food science, 2006, 43, 113-116.
Swiegers, J. H., Capone, D. L., Pardon, K. H., Elsey, G. M., Sefton, M. A., Francis, I. L., y Pretorius, I. S.: “Engineering volatile thiol release in Saccharomyces cerevisiae for improved wine aroma”. Yeast, 2007, 24(7), 561-574.
Thibon, C., Marullo, P., Claisse, O., Cullin, C., Dubourdieu, D., y Tominaga, T.: “Nitrogen catabolic repression controls the release of volatile thiols by Saccharomyces cerevisiae during wine fermentation”. FEMS Yeast Research, 2008, 8(7), 1076–1086.
Thibon, C., Cluzet, S., Mérillon, J. M., Darriet, P., y Dubourdieu, D.: “3-sulfanylhexanol precursor biogenesis in grapevine cells: the stimulating effect of Botrytis cinerea”. Journal of Agricultural and Food Chemistry, 2011, 59, 1344–1351.
Tofalo, R., Perpetuini, G., Battistelli, N., Tittarelli, F., y Suzzi G.: “Correlation between IRC7 gene expression and 4-mercapto-4-methylpentan-2-one production in Saccharomyces cerevisiae strains”. Yeast, 2020, 37, 487–495.
Vicente, J., Kiene, F., Fracassetti, D., De Noni, I., Shemehen, R., Tarasov, A., Dobrydnev, AV., Marquina, D., Santos, A., Rauhut, D., Belda, I., y Ruiz, J.: “Precursors consumption preferences and thiol release capacity of the wine yeasts Saccharomyces cerevisiae, Torulaspora delbrueckii, and Lachancea thermotolerans”. International Journal of Food Microbiology, 2024, 425, 110858.
Walker, R. S., y Pretorius, I. S.: “Synthetic biology for the engineering of complex wine yeast communities”. Nature Food, 2022, 3(4), 249-254.
Zott, K., Thibon, C., Bely, M., Lonvaud-Funel, A., Dubourdieu, D., y Masneuf-Pomarede, I.: “The grape must non-Saccharomyces microbial community: impact on volatile thiol release”. International Journal of Food Microbiology, 2011, 151(2), 210-215.